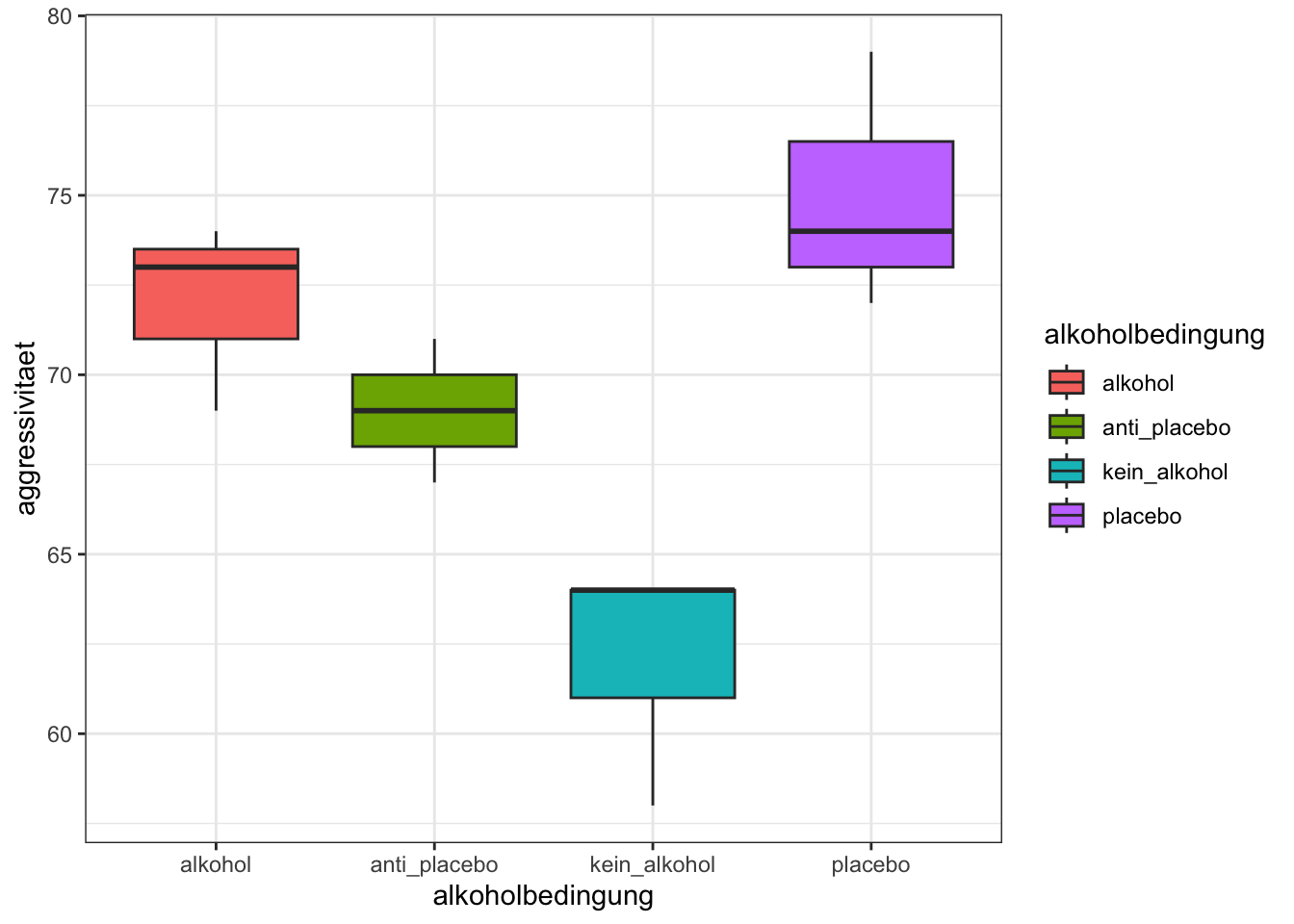
kein_alkohol <- c(64, 58, 64)
placebo <- c(74, 79, 72)
anti_placebo <- c(71, 69, 67)
alkohol <- c(69, 73, 74)3 Datensätze
Wir werden nun anfangen, mit Datensätzen als Data Frames zu arbeiten. Zunächst werden wir diese selber kreieren, und dann Datensätze aus verschiedenen Datenformaten importieren.
3.1 Datensätze selber erstellen
3.1.1 Ohne Messwiederholung
Wir erstellen nun einen Datensatz mit einem between-subjects Faktor.
Wie können wir aus vier einzelnen Vektoren einen solchen Data Frame erstellen? Eine mögliche Lösung ist, zunächst aus jedem der vier Vektoren einen Data Frame zu erstellen und die Alkoholbedingung jeweils als weitere Variable hinzuzufügen. Anschliessend können wir die vier Data Frames zu einem zusammenfügen, und die Alkoholbedingung zu einem factor konvertieren.
Wir wiederholen hier die Erstellung der einzelnen Vektoren, damit es noch klarer wird:
# Zuerst dplyr package laden:
library(dplyr)kein_alkohol <- c(64, 58, 64)
kein_alkohol <- tibble(
aggressivitaet = kein_alkohol,
alkoholbedingung = "kein_alkohol"
)
kein_alkohol# A tibble: 3 × 2
aggressivitaet alkoholbedingung
<dbl> <chr>
1 64 kein_alkohol
2 58 kein_alkohol
3 64 kein_alkohol placebo <- c(74, 79, 72)
placebo <- tibble(
aggressivitaet = placebo,
alkoholbedingung = "placebo"
)
placebo# A tibble: 3 × 2
aggressivitaet alkoholbedingung
<dbl> <chr>
1 74 placebo
2 79 placebo
3 72 placebo anti_placebo <- c(71, 69, 67)
anti_placebo <- tibble(
aggressivitaet = anti_placebo,
alkoholbedingung = "anti_placebo"
)
anti_placebo# A tibble: 3 × 2
aggressivitaet alkoholbedingung
<dbl> <chr>
1 71 anti_placebo
2 69 anti_placebo
3 67 anti_placebo alkohol <- c(69, 73, 74)
alkohol <- tibble(
aggressivitaet = alkohol,
alkoholbedingung = "alkohol"
)
alkohol# A tibble: 3 × 2
aggressivitaet alkoholbedingung
<dbl> <chr>
1 69 alkohol
2 73 alkohol
3 74 alkohol Nun können wir diese vier Data Frames mit rbind() oder bind_rows() zusammenfügen. Die Funktion bind_rows() ist im dplyr package, und hat einige Vorteile (in diesem Beispiel spielt es keine Rolle, welche Sie benützen).
alk_aggr <- bind_rows(
kein_alkohol,
placebo,
anti_placebo,
alkohol
)alk_aggr# A tibble: 12 × 2
aggressivitaet alkoholbedingung
<dbl> <chr>
1 64 kein_alkohol
2 58 kein_alkohol
3 64 kein_alkohol
4 74 placebo
5 79 placebo
6 72 placebo
7 71 anti_placebo
8 69 anti_placebo
9 67 anti_placebo
10 69 alkohol
11 73 alkohol
12 74 alkohol Nun müssen wir noch die Variable alkoholbedingung zu einem factor konvertieren:
alk_aggr$alkoholbedingung <- factor(alk_aggr$alkoholbedingung)alk_aggr# A tibble: 12 × 2
aggressivitaet alkoholbedingung
<dbl> <fct>
1 64 kein_alkohol
2 58 kein_alkohol
3 64 kein_alkohol
4 74 placebo
5 79 placebo
6 72 placebo
7 71 anti_placebo
8 69 anti_placebo
9 67 anti_placebo
10 69 alkohol
11 73 alkohol
12 74 alkohol Der Datensatz ist nun komplett, wir wollen aber noch die Variablenreihenfolge umkehren:
alk_aggr <- alk_aggr[2:1]
alk_aggr# A tibble: 12 × 2
alkoholbedingung aggressivitaet
<fct> <dbl>
1 kein_alkohol 64
2 kein_alkohol 58
3 kein_alkohol 64
4 placebo 74
5 placebo 79
6 placebo 72
7 anti_placebo 71
8 anti_placebo 69
9 anti_placebo 67
10 alkohol 69
11 alkohol 73
12 alkohol 74In diesem Datensatz haben wir zwei Variablen - einen Gruppierungsfaktor alkoholbedingung und eine Messvariable aggressivitaet, und die Stufen des Faktors sind nicht messwiederholt. Jede Beobachtung steht auf einer Zeile, und jede Variable steht in einer Spalte - ein solcher Datensatz ist im long Format dargestellt.
3.1.2 Mit Messwiederholung
Bei einem Faktor mit Messwiederholung sind wir oft von anderen Statistikprogrammen wie SPSS oder jamovi ein anderes Format gewöhnt. Wenn die Stufen des repeated measures Faktors nicht in einer Spalte stehen, sondern in separaten Spalten, ist der Datensatz nicht im long, sondern im wide Format. Wir veranschaulichen das mit einem weiteren Beispiel.
Wir konstruieren nun diesen Datensatz im wide Format.
vpn <- c("vp_1", "vp_2", "vp_3", "vp_4")
vpn <- factor(vpn)
# n ist die Anzahl vpn
n <- length(vpn)# Daten werden simuliert - Statistiker:innen machen so etwas oft, um etwas zu
# illustrieren oder um statistische Verfahren zu testen.
set.seed(1234)
zufriedenheit_t1 <- round(rnorm(n, mean = 60, sd = 10), digits = 2)
zufriedenheit_t1[1] 47.93 62.77 70.84 36.54zufriedenheit_t2 <- round(rnorm(n, mean = 75, sd = 10), digits = 2)
zufriedenheit_t2[1] 79.29 80.06 69.25 69.53Nun können wir die Variablen mit tibble() zu einem Datensatz zusammenfügen:
zufriedenheit <- tibble(
vpn = vpn,
zufriedenheit_t1 = zufriedenheit_t1,
zufriedenheit_t2 = zufriedenheit_t2
)
zufriedenheit# A tibble: 4 × 3
vpn zufriedenheit_t1 zufriedenheit_t2
<fct> <dbl> <dbl>
1 vp_1 47.9 79.3
2 vp_2 62.8 80.1
3 vp_3 70.8 69.2
4 vp_4 36.5 69.5Mit diesem Datensatz können wir in R z.B. einen t-Test durchführen, das wide Format ist aber für viele Anwendungen nicht optimal, denn viele statistische Verfahren verlangen einen Datensatz im long Format. Um die Daten mit ggplot2 grafisch darzustellen, ist es ebenfalls sinnvoll, den Datensatz in ein long Format zu konvertieren. Datenkonvertierung wird oft als Reshaping bezeichnet. Wir werden im nächsten Kapitel genauer darauf eingehen, wenn wir die tidyverse Packages kennenlernen. Ein Begriff, welchen wir immer häufiger antreffen, ist tidy Data. Dies bezieht sich eben auf das Datenformat: das long Format wird bevorzugt (ist tidier als das wide Format). Wenn wir uns daran halten, erleichtern wir uns die Arbeit mit R erheblich, und wir können von den tidyverse Packages profitieren, welche alle sehr gut aufeinander abgestimmt sind.
Bevor wir das Data Reshaping automatisieren, ist es aber sinnvoll, einmal einen Datensatz manuell von wide zu long zu konvertieren.
3.1.3 Manuelle Konversion von wide zu long
Jede Person gibt zu zwei Messzeitpunkten ein Zufriedenheits-Rating - diese Ratings stellen die Beobachtungen dar. In einem long Datensatz stehen die Beobachtungen in den Zeilen, daher erhält jede Beobachtung eine Zeile, und nicht mehr jede Person (da jede Person jetzt zwei Beobachtungen hat, die den Zeitpunkten der Erhebung entsprechen). Die Werte in der Personen/ID-Variable (hier: vpn) werden gemäss der Anzahl Beobachtungen pro Person dupliziert.
# Wir nennen den Datensatz nun zufriedenheit_wide
zufriedenheit_wide <- zufriedenheitvpn <- rep(zufriedenheit_wide$vpn, each = 2)
vpn[1] vp_1 vp_1 vp_2 vp_2 vp_3 vp_3 vp_4 vp_4
Levels: vp_1 vp_2 vp_3 vp_4Nun müssen wir aus den beiden Variablen zufriedenheit_t1 und zufriedenheit_t2 des wide Datensatzes einen Faktor messzeitpunkt und eine Variable zufriedenheit erstellen:
messzeitpunkt <- rep(c("t1", "t2"), times = n)
messzeitpunkt <- as.factor(messzeitpunkt)
messzeitpunkt[1] t1 t2 t1 t2 t1 t2 t1 t2
Levels: t1 t2zufriedenheit <- c(rbind(
zufriedenheit_wide$zufriedenheit_t1,
zufriedenheit_wide$zufriedenheit_t2
))
# oder
zufriedenheit <- as.vector(rbind(
zufriedenheit_wide$zufriedenheit_t1,
zufriedenheit_wide$zufriedenheit_t2
))
zufriedenheit[1] 47.93 79.29 62.77 80.06 70.84 69.25 36.54 69.53Wir haben nun alle drei Variablen, die wir für den Datensatz im long Format brauchen, und können diese nun zusammenfügen:
zufriedenheit_long <- tibble(
vpn = vpn,
messzeitpunkt = messzeitpunkt,
zufriedenheit = zufriedenheit
)zufriedenheit_long# A tibble: 8 × 3
vpn messzeitpunkt zufriedenheit
<fct> <fct> <dbl>
1 vp_1 t1 47.9
2 vp_1 t2 79.3
3 vp_2 t1 62.8
4 vp_2 t2 80.1
5 vp_3 t1 70.8
6 vp_3 t2 69.2
7 vp_4 t1 36.5
8 vp_4 t2 69.5Der Datensatz mit Messwiederholung kann jetzt ähnlich wie derjenige ohne Messwiederholung mit ggplot2 grafisch dargestellt werden.
library(ggplot2)
zufriedenheit_long |>
ggplot(aes(
x = messzeitpunkt,
y = zufriedenheit,
group = vpn, colour = vpn
)) +
geom_point(size = 4) +
geom_line(size = 2) +
theme_bw() # macht den Hintergrund weiss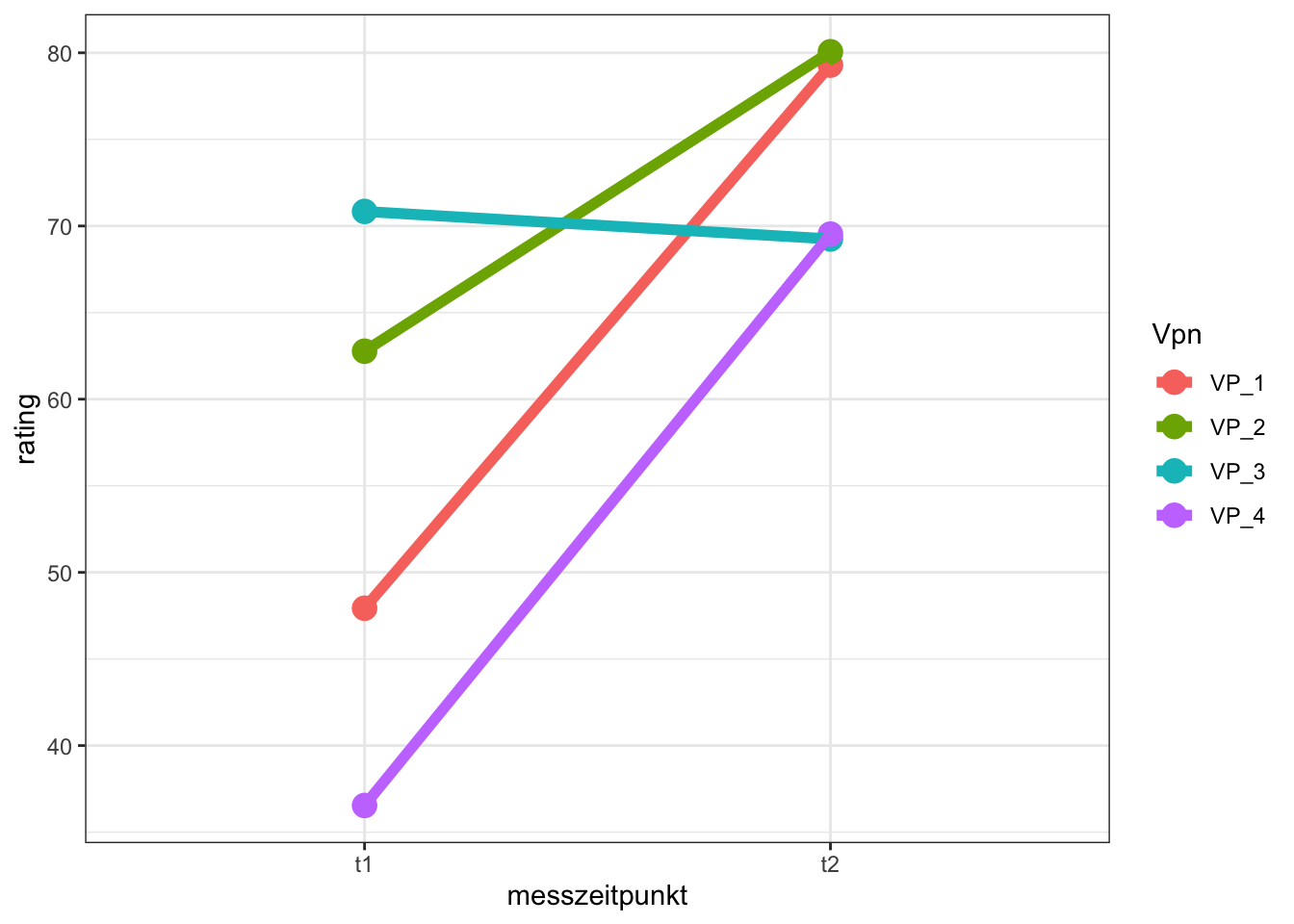
Manuelles Reshaping ist eher mühsam, und sollte vermieden werden. Das obige Beispiel soll nur zur Illustration dienen. Wir werden in Zukunft dafür eine Funktion aus dem tidyr Package verwenden: pivot_longer().
Als kleine Vorschau: die Konvertierung von wide zu long würden wir so machen:
library(tidyr)
library(stringr)
zufriedenheit_wide# A tibble: 4 × 3
vpn zufriedenheit_t1 zufriedenheit_t2
<fct> <dbl> <dbl>
1 vp_1 47.9 79.3
2 vp_2 62.8 80.1
3 vp_3 70.8 69.2
4 vp_4 36.5 69.5zufriedenheit_long <- zufriedenheit_wide |>
pivot_longer(c(zufriedenheit_t1, zufriedenheit_t2),
names_to = "messzeitpunkt",
values_to = "zufriedenheit")
zufriedenheit_long# A tibble: 8 × 3
vpn messzeitpunkt zufriedenheit
<fct> <chr> <dbl>
1 vp_1 zufriedenheit_t1 47.9
2 vp_1 zufriedenheit_t2 79.3
3 vp_2 zufriedenheit_t1 62.8
4 vp_2 zufriedenheit_t2 80.1
5 vp_3 zufriedenheit_t1 70.8
6 vp_3 zufriedenheit_t2 69.2
7 vp_4 zufriedenheit_t1 36.5
8 vp_4 zufriedenheit_t2 69.5Jetzt wollen wir noch zufriedenheit_ von den Faktorstufen entfernen, so dass die Stufen t1 und t2 heissen. Dafür verwenden wir die Funktion str_replace().
zufriedenheit_long$messzeitpunkt <- zufriedenheit_long$messzeitpunkt |>
str_replace(".*_", "") |>
as.factor()
zufriedenheit_long# A tibble: 8 × 3
vpn messzeitpunkt zufriedenheit
<fct> <fct> <dbl>
1 vp_1 t1 47.9
2 vp_1 t2 79.3
3 vp_2 t1 62.8
4 vp_2 t2 80.1
5 vp_3 t1 70.8
6 vp_3 t2 69.2
7 vp_4 t1 36.5
8 vp_4 t2 69.5Die obige Schreibweise benützt den Pipe Operator |>: wir werden bald mehr darüber erfahren. Äquivalent dazu können wir die Funktionen auch verschachtelt aufrufen:
zufriedenheit_long <- pivot_longer(c(zufriedenheit_t1, zufriedenheit_t2),
names_to = "messzeitpunkt",
values_to = "zufriedenheit"
)
zufriedenheit_long$messzeitpunkt <-
as.factor(str_replace(
zufriedenheit_long$messzeitpunkt,
".*_", ""
))3.1.4 Long vs. wide
Um den Unterschied zwischen einem long und einem wide Datensatz zu verdeutlichen, zeigen wir hier nochmals beide Varianten:
# wide
zufriedenheit_wide# A tibble: 4 × 3
vpn zufriedenheit_t1 zufriedenheit_t2
<fct> <dbl> <dbl>
1 vp_1 47.9 79.3
2 vp_2 62.8 80.1
3 vp_3 70.8 69.2
4 vp_4 36.5 69.5# long
zufriedenheit_long# A tibble: 8 × 3
vpn messzeitpunkt zufriedenheit
<fct> <fct> <dbl>
1 vp_1 t1 47.9
2 vp_1 t2 79.3
3 vp_2 t1 62.8
4 vp_2 t2 80.1
5 vp_3 t1 70.8
6 vp_3 t2 69.2
7 vp_4 t1 36.5
8 vp_4 t2 69.53.2 Daten importieren
Meistens arbeiten wir jedoch nicht mit selber generierten Datensätzen, sondern wollen diese aus Textdateien, Excel-Spreadsheets oder SPSS-Dateien importieren.
Laden Sie bitte folgende Datensätze von GitHub herunter, indem Sie Rechtsklick auf die folgenden Datensätze machen und dann “Ziel speichern unter” wählen:
Es handelt sich um den Datensatz zur Zufriedenheit, den wir oben erstellt haben. zufriedenheit.csv ist eine Textdatei, in dem die Spalten durch Kommata getrennt sind (csv = comma-separated values). zufriedenheit-semicolon.csv ist ebenfalls eine Textdatei, die Spalten sind hier aber durch Strichpunkte (Semikolons) getrennt. zufriedenheit.sav und zufriedenheit.xls sind SPSS- bzw. Excel-Dateien.
Kreieren Sie in Ihrem Projektordner in RStudio einen neuen Ordner und nennen Sie diesen data. Speichern Sie nun die heruntergeladenen Datenfiles in diesem Ordner. Wir werden diese Dateien nun der Reihe nach importieren.
Es gibt in RStudio zwei Möglichkeiten, Datensätze zu importieren:
Mit Funktionsaufrufen:
read_csv()bzw.read_delim()(für ‘;’),read_sav()undread_excel().Mit dem GUI: Das Menu kann entweder via ‘File > Import Dataset’ oder im Environment-Bereich aufgerufen werden.
Die zweite Option ist am Anfang einfacher und hat zwei Vorteile. Erstens werden alle Optionen für den Import angezeigt (diese sind auch als Funktionsargumente verfügbar), und zweitens generiert RStudio den entsprechenden R Code. Wenn man sich nicht sicher ist, kann man beim ersten Mal das GUI benützen, und in Zukunft dann den generierten Code. Dies ist oft schneller und hat wiederum den Vorteil, dass man alle Schritte für die Datenanalyse in einem R Script/Notebook festhalten kann.
3.2.1 CSV-Dateien
Die Funktion, welche wir benötigen, um CSV-Dateien zu importieren, befindet sich im readr Package. Dieses müssen wir zuerst laden:
library(readr)Da dieses Package zu dem Metapackage tidyverse gehört, können wir einfach dieses laden:
library(tidyverse)Zuerst importieren wir den Datensatz via GUI. Klicken Sie in Environment auf Import Dataset > From Text (readr) (oder ‘File > Import Dataset > From Text (readr)’). Im Dialogfenster sehen Sie rechts unten einen Code Preview. Hier steht am Anfang:
library(readr)
dataset <- read_csv(NULL)
View(dataset)Links unten sehen Sie alle Optionen für den Import. Diese Optionen sind Argumente der read_csv() oder der allgemeineren read_delim() Funktion:
args(read_csv)function (file, col_names = TRUE, col_types = NULL, col_select = NULL,
id = NULL, locale = default_locale(), na = c("", "NA"), quoted_na = TRUE,
quote = "\"", comment = "", trim_ws = TRUE, skip = 0, n_max = Inf,
guess_max = min(1000, n_max), name_repair = "unique", num_threads = readr_threads(),
progress = show_progress(), show_col_types = should_show_types(),
skip_empty_rows = TRUE, lazy = should_read_lazy())
NULLWählen Sie über File / Browse die Datei zufriedenheit.csv aus. Sie sehen nun den Pfad dieser Datei sowie eine Data Preview. Hier werden die Daten gezeigt, mit Angaben zum Datentyp.
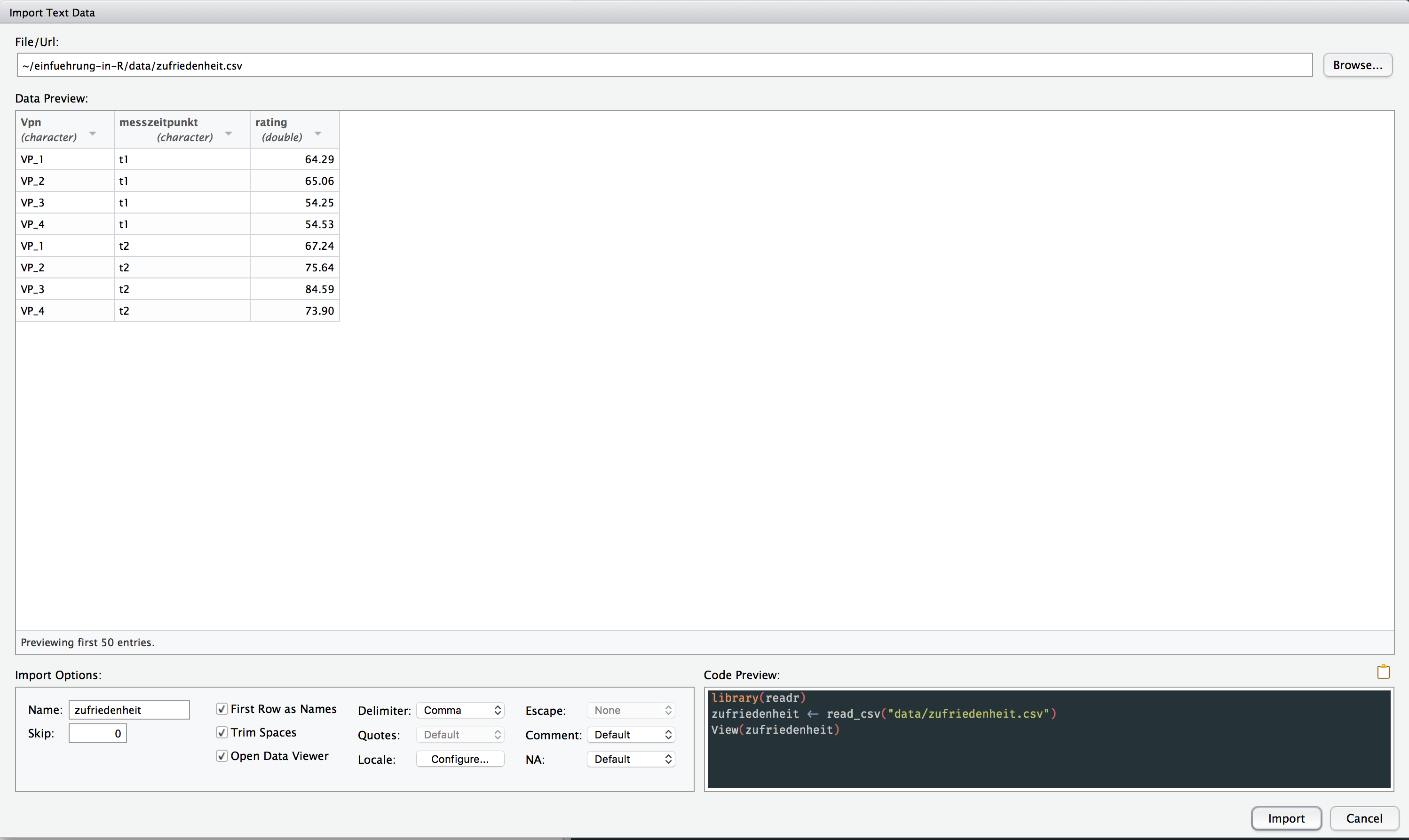 \(~\)
\(~\)
Sie sollten sehen, dass sowohl die Variable vpn als auch die Variable messzeitpunkt als character vectors importiert wurden. Diese werden wir nachher zu Faktoren konvertieren.
Bei Import Options wird automatisch ein Name für den Data Frame generiert; dieser entspricht dem Dateinamen (ohne das Suffix ‘.csv.’).
Sie probieren am besten selbst aus, welche Auswirkung es hat, wenn Sie die Optionen ändern. Wenn Sie z.B. “First Row as Names” nicht auswählen, wird R die Variablennamen, die in der ersten Zeile der CSV-Datei stehen, nicht verwenden. Eine weitere wichtige Option ist “NA”: hier können Sie angeben, welche Werte in der Datei als fehlende Werte behandelt werden sollen.
Nun klicken Sie bitte auf Import. Im Environment-Bereich erscheint ein Data Frame (zufriedenheit), und der Code, der verwendet wurde, erscheint in der Konsole.
library(readr)
zufriedenheit <- read_csv("data/zufriedenheit.csv")Rows: 8 Columns: 3
── Column specification ────────────────────────────────────────────────────────
Delimiter: ","
chr (2): vpn, messzeitpunkt
dbl (1): zufriedenheit
ℹ Use `spec()` to retrieve the full column specification for this data.
ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.Nun müssen wir die beiden Gruppierungsvariablen zu Faktoren konvertieren:
zufriedenheit$vpn <- as.factor(zufriedenheit$vpn)
zufriedenheit$messzeitpunkt <- as.factor(zufriedenheit$messzeitpunkt)zufriedenheit# A tibble: 8 × 3
vpn messzeitpunkt zufriedenheit
<fct> <fct> <dbl>
1 vp_1 t1 64.3
2 vp_2 t1 65.1
3 vp_3 t1 54.2
4 vp_4 t1 54.5
5 vp_1 t2 67.2
6 vp_2 t2 75.6
7 vp_3 t2 84.6
8 vp_4 t2 73.9\(~\)
Alternativ können Variablen auch bereits beim Einlesen via GUI formatiert werden. Öffnen sie dazu nochmals via Import Dataset > From Text (readr) > Browse den Datensatz zufriedenheit.csv. Durch das Klicken auf einen der Spaltennamen im Data Preview öffnet sich ein Dropdown:
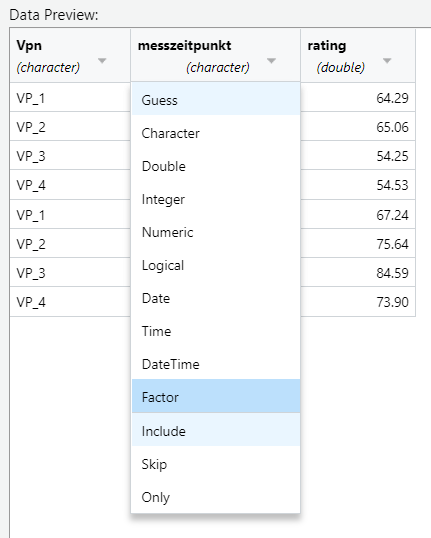
Dieses Dropdown hat zwei Teile: im ersten Teil (von Guess bis Factor) können sie wählen, welches Format die Spalte haben soll (standardmässig auf Guess). Im zweiten Teil (Include bis Only) kann gesteuert werden, ob die Spalte eingelesen werden soll oder nicht (standardmässig auf Include). Wir wählen hier Factor, um die Spalte als Faktor einzulesen. Dies öffnet ein weiteres Fenster. Hier geben wir die gewünschten Faktorstufen ein, jeweils getrennt durch ein Komma:
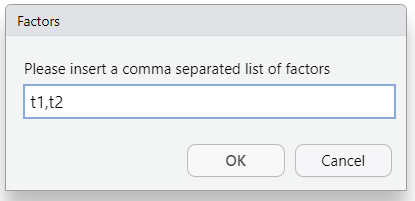
Im Code Preview werden diese Änderungen nun ebenfalls angezeigt. Wenn Sie den Datensatz das nächste Mal einlesen, werden diese Einstellungen also direkt übernommen.
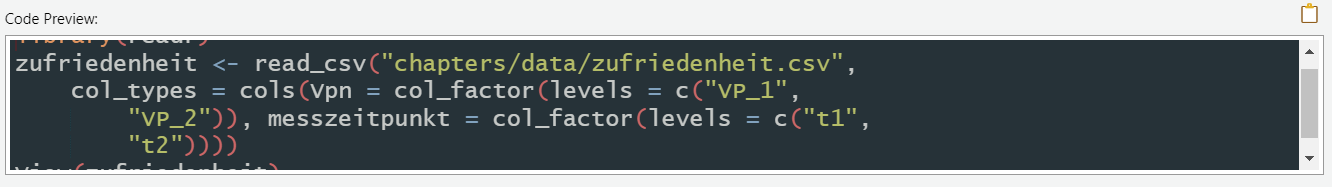
Diese Methode der Faktor-Konvertierung ist am Anfang deutlich einfacher als die manuelle Methode mit as.factor() oder factor(). Es ist jedoch wichtig, sich auch mit letzteren vertraut zu machen, da auch nach Einlesen eines Datensatzes häufig damit gearbeitet werden muss, z.B. wenn eine (weitere) Faktorvariable aus einer bestehenden Variable erstellt werden soll usw..
Wenn wir einen Data Frame als CSV-Datei speichern wollen, verwenden wir dafür die Funktion write_csv():
write_csv(x = zufriedenheit, file = "data/zufriedenheit.csv")3.2.2 SPSS-Dateien
Nun importieren wir denselben Datensatz, aber dieses Mal von einer SPSS-Datei (zufriedenheit.sav). Dafür benötigen wir die Funktion read_sav(). Diese befindet sich im Package haven, welches wir zuerst noch installieren müssen:
install.packages("haven")Sobald haven installiert ist, können Sie auch im Environment auf Import Dataset > From SPSS klicken und die Datei auswählen. Sie sehen dann den Code Preview:
library(haven)
zufriedenheit_spss <- read_sav("data/zufriedenheit.sav")Im Gegensatz zum Importieren von CSV-Dateien gibt es hier keine Optionen - mit Ausnahme des Namens, den der Data Frame nach dem Import erhält. Diesen ändern wir vom vorgeschlagenen zufriedenheit zu zufriedenheit_spss (um den vorher importierten Data Frame nicht zu überschreiben). Klicken Sie auf Import, der Data Frame zufriedenheit_spss erscheint anschliessend im Global Environment.
Im Unterschied zu dem von einer CSV-Datei importierten Data Frame haben die Variablen zufriedenheit_spss weitere Attribute. Das wichtigste dabei ist das Labels Attribut:
zufriedenheit_spss$vpn<labelled<double>[8]>
[1] 1 2 3 4 1 2 3 4
Labels:
value label
1 vp_1
2 vp_2
3 vp_3
4 vp_4Dieses beinhaltet die Wertelabels aus einem SPSS-Datensatz. In SPSS, einem weit verbreiteten kommerziellen Statistikprogramm, werden - anders als in R - auch Faktorstufen (also Gruppen- oder Kategorienbezeichnungen) grundsätzlich numerisch kodiert (z.B. für Geschlecht: 0 für maennlich, 1 für weiblich und 2 für divers - oder beliebige andere Zahlen). Die eigentlichen Bezeichnungen der Faktorstufen werden dann zusätzlich als Attribute der Werte hinterlegt (sogenannte Wertelabels).
Die so aus einem SPSS-Datensatz eingelesene Variable ist zunächst ein Hybrid aus einer numerischen und einer Faktor-Variable, eine so genannte labelled double Variable. In unserem Beispiel werden die Werte der Versuchspersonen-Variable vpn als numerische Werte eingelesen, die aber jeweils ein Label besitzen, das die eigentliche Bezeichnung der Faktorstufe (hier des Personen-Faktors) darstellt.
Um die als labelled double eingelesenen Variablen zu Faktoren konvertieren, muss nach dem Einlesen der Daten die Funktion as_factor() aus haven verwendet werden. Diese Funktion erlaubt es, entweder die numerischen Werte oder die Wertelabels (oder beide zusammengefügt) als Stufen des zu erstellenden Faktors zu definieren. Die Einstellung erfolgt mit dem Argument levels. Die für uns sinnvollste Einstellung ist in den meisten Fällen levels = "default". Damit werden die Wertelabels als Faktorstufen benützt, sofern solche vorhanden sind, andernfalls wird auf die Werte selbst zurückgegriffen. "default" ist zudem auch die Standardeinstellung, sodass beim Weglassen des levels-Arguments standardmässig die Wertelabels als Faktorstufen eingelesen werden. Die anderen levels-Optionen sind "both" (Werte und Wertelabels werden zusammengefügt), "labels" (nur Labels, NA falls keine vorhanden) und "values" (nur Werte).
Argumente der Funktion as_factor()
levels
How to create the levels of the generated factor:
"default": uses labels where available, otherwise the values. Labels are sorted by value.
"both": like "default", but pastes together the level and value
"labels": use only the labels; unlabelled values become NA
"values": use only the values
ordered
If TRUE create an ordered (ordinal) factor, if FALSE (the default) create a regular (nominal) factor.Mit dem Argument ordered, welches entweder TRUE oder FALSE (Standardeinstellung) ist, kann ein ordinaler Faktor erstellt werden, d.h. ein Faktor, dessen Stufen in einer Rangfolge stehen. In den meisten Fällen wird diese Einstellung nicht benötigt, das Argument kann daher normalerweise weggelassen werden (default: ordered = FALSE).
zufriedenheit_spss$vpn <- as_factor(zufriedenheit_spss$vpn,
levels = "default"
)
zufriedenheit_spss$messzeitpunkt <- as_factor(zufriedenheit_spss$messzeitpunkt,
levels = "default"
)So können R Data Frames als SPSS-Datendatei gespeichert werden (Dateiendung: .sav):
write_sav(data = zufriedenheit_spss, path = "data/zufriedenheit.sav")3.2.3 Excel-Dateien
Auch Excel-Dateien können importiert werden. Wählen Sie Import Dataset > From Excel, und wählen Sie dann die heruntergeladene Excel-Datei zufriedenheit.xlsx aus. Nennen Sie den Data Frame zufriedenheit_xl (um ihn von den bereits importierten Data Frames zu unterscheiden). Unter dem Name-Feld sehen sie ein Dropdown-Menu mit dem Namen Sheet. Hier können Sie angeben, welches Worksheet der Excel-Datei Sie importieren möchten. Wenn Sie hier zufriedenheit auswählen, erscheint folgender R Code im Code Preview:
library(readxl)
zufriedenheit_xl <- read_excel("data/zufriedenheit.xlsx",
sheet = "zufriedenheit"
)Die benötigte Funktion heisst read_excel() und befindet sich im readxl package. Dieses muss zuvor explizit geladen werden, d.h. es wird nicht automatisch mit dem Befehl library(tidyverse) geladen.
Auch hier müssen kategoriale Variablen anschliessend zu Faktoren konvertiert werden:
zufriedenheit_xl$vpn <- as.factor(zufriedenheit_xl$vpn)
zufriedenheit_xl$messzeitpunkt <- as.factor(zufriedenheit_xl$messzeitpunkt)3.2.4 RData-Dateien
Unsere letzte Option ist eine RData-Datei. Dies ist ein binary- Datei, d.h. es handelt sich in diesem Fall um keine Textdatei. Wir können mehrere Objekte aus dem R Workspace in eine RData-Datei speichern, und wieder daraus laden. Der grosse Vorteil daran ist, dass Attribute, wie z.B. die Objektklasse factor und die Faktorstufen levels mitgespeichert werden, und nicht wie beim Speichern z.B. als CSV- oder Excel-Datei verloren gehen. Ein weiterer Vorteil ist, dass die Datei komprimiert werden kann. Demgegenüber steht die Tatsache, dass eine RData Datei nicht ohne Weiteres von anderen Statistikprogrammen gelesen werden kann. Deshalb sollte man besonders dann Textdateien (.csv) verwenden, wenn man einen Datensatz anderen Personen zur Verfügung stellen will.
Ausserdem ist es empfehlenswert, alle Schritte der Datenanalyse (inkl. Einlesen der Daten) in einem R Script File festzuhalten. Der Import von Dateien und die Umwandlung von Variablen kann so nachvollzogen und das Einlesen der Daten bei späteren R Sitzungen automatisiert werden.
Mit der Funktion save() können wir Objekte, die sich im Global Environment befinden, direkt als .RData (oder .Rda) Dateien speichern. Z.B. können wir so den Data Frame zufriedenheit als RData-Datendatei auf unserem Computer speichern:
save(zufriedenheit, file = "data/zufriedenheit.Rda")Wir können dieser Funktion auch eine Liste von Objekten als Argument übergeben:
save(zufriedenheit, zufriedenheit_spss, zufriedenheit_xl,
file = "data/zufriedenheit_alle.Rda"
)zufriedenheit_alle.Rda enthält nun alle drei Data Frames.
Mit load() können wir diese auch wieder laden:
load(file = "data/zufriedenheit_alle.Rda")