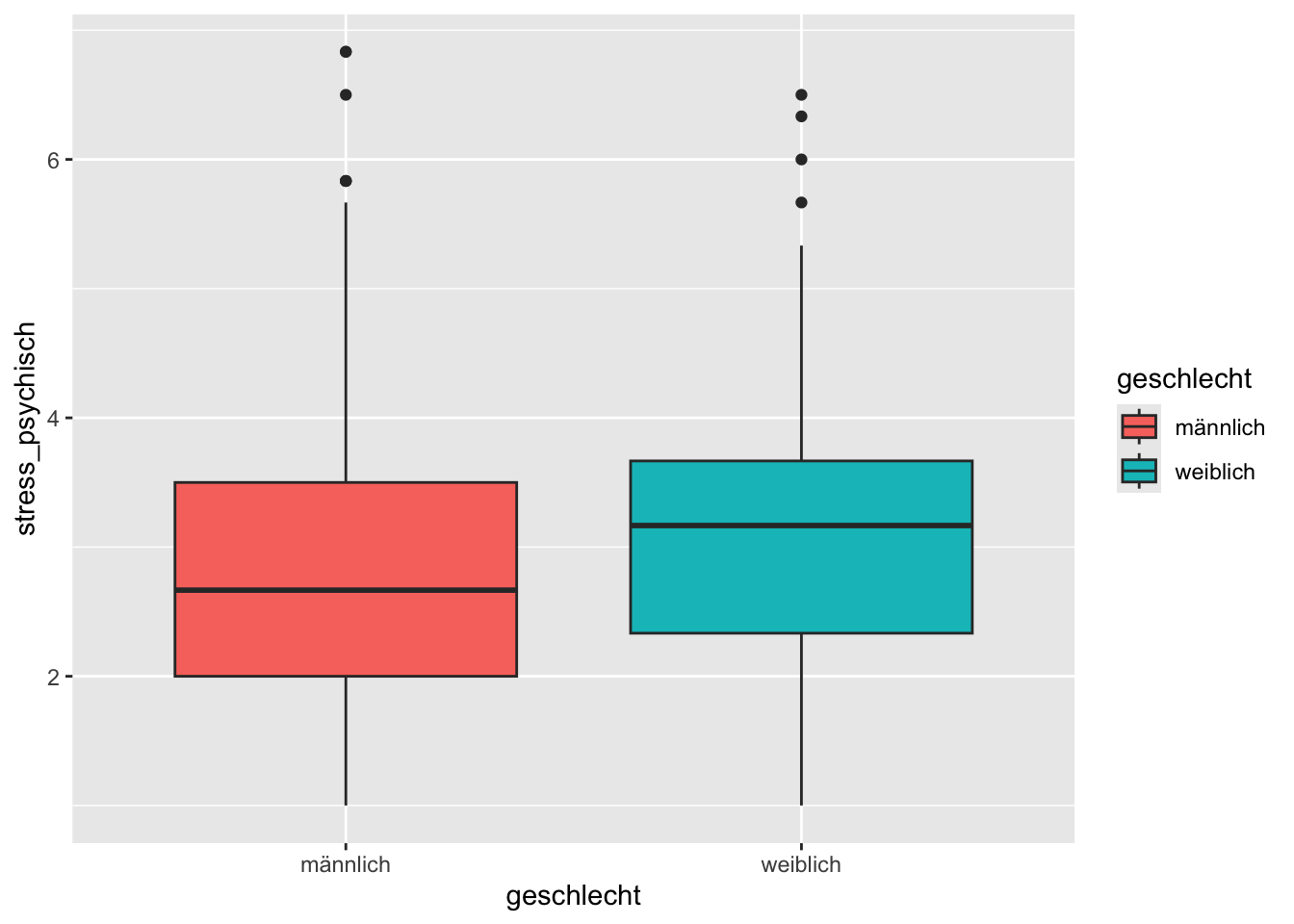
# A tibble: 2 × 4
name t1 t2 t3
<fct> <dbl> <dbl> <dbl>
1 Marshall 4 5 7
2 Skye 3 6 74 Daten transformieren
4.1 Tidy data
Wenn in der Statistik/Data Science über Datensätze gesprochen wird, werden oft die Begriffe wide und long verwendet. Diese beziehen sich auf die Form eines Datensatzes: ein wide Datensatz hat mehr Spalten als ein entsprechender long Datensatz und umgekehrt hat ein long Datensatz mehr Zeilen als ein entsprechender wide Datensatz.
Genauer gesagt hat in einem long Datensatz jede Beobachtung eine eigene Zeile und jede Variable ist eine Spalte. In einem wide Datensatz können (insbesondere messwiederholte) Variablen über mehrere Spalten verteilt werden, und somit jede Zeile eine bestimmte Person repräsentieren.
Betrachten wir folgendes Beispiel: Wir haben zwei Beobachtungen (irgendeiner psychologischen Variable) für zwei Personen, zu drei verschiedenen Zeitpunkten. Nennen wir die Zeitpunkte t1, t2 und t3. Die folgende Darstellung eines solchen Datensatzes gilt als wide format:
Hier steht jede Zeile für eine Person und die Beobachtungen sind auf die drei Spalten t1, t2 und t3 verteilt. Diese Spalten repräsentieren aber eigentlich die drei Stufen eines (messwiederholten) Faktors zeit. Wir können genau die gleichen Daten in einem längeren Format darstellen, in dem jede Zeile einer Beobachtung entspricht und jede Spalte eine Variable darstellt. Insgesamt gibt es dann drei Variablen: name, zeit und punkte, wobei punkte hier als Platzhalter für die gemessenene (psychologische) Variable steht, deren Ausprägungen in dieser Spalte aufgeführt werden.
Um den obigen wide Datensatz (als df gespeichert) in einen long Datensatz zu transformieren, verwenden wir pivot_longer() aus dem package tidyr. Die Details zu dieser Funktion behandeln wir weiter unten im Abschnitt Reshaping: tidyr.
df |>
pivot_longer(!name, names_to = "zeit", values_to = "punkte")# A tibble: 6 × 3
name zeit punkte
<fct> <chr> <dbl>
1 Marshall t1 4
2 Marshall t2 5
3 Marshall t3 7
4 Skye t1 3
5 Skye t2 6
6 Skye t3 7Wir haben jetzt nicht mehr für jede Person eine Zeile. Die Werte von Variablen, die sich nicht für jede Beobachtung (= jede Zeile) ändern, werden jetzt über die Zeilen hinweg wiederholt (gilt für name und zeit).
Ein Datensatz, der auf diese Weise organisiert ist, wird oft auch als tidy bezeichnet.
Wie wir später noch sehen werden, erfordern viele Arten von statistischen Analysen und insbesondere Grafik-Funktionen einen long Datensatz, und daher muss oft erstaunlich viel Zeit für die Organisation von Daten für die weitere Analyse aufgewendet werden (diese Art von Arbeit wird oft als “data wrangling” bezeichnet).
In diesem Kurs werden wir mit den tidyverse Packages zur Manipulation und Transformation von Daten arbeiten. Natürlich gibt es auch andere Möglichkeiten der Datenbearbeitung, aber unserer Ansicht nach bietet die tidyverse-Methode die konsistenteste Art der Arbeit mit Daten und reduziert daneben auch die kognitive Belastung der Benutzer.
Die Pakete, die für Datenmanipulationen verwendet werden, sind tidyr für die Transformation/Umformung von Datensätzen und dplyr für die Manipulation/Bearbeitung von Datensätzen und den dort enthaltenen Variablen. Für Faktoren benutzen wir zudem eine Funktion aus dem package forcats. Die Funktionen, die wir uns anschauen werden, sind:
| Package | Funktion | Verwendung |
|---|---|---|
tidyr |
pivot_longer() |
erhöht die Anzahl der Zeilen, verringert die Anzahl der Spalten |
tidyr |
pivot_wider() |
verringert die Anzahl der Zeilen, erhöht die Anzahl der Spalten |
tidyr |
drop_na() |
löscht alle Zeilen eines Datensatzes, die missing values (NA) enthalten |
dplyr |
rename() |
zum Umbenennen von Variablen |
dplyr |
select() |
wählt Variablen (Spalten) aus |
dplyr |
relocate() |
verändert die Reihenfolge von Variablen (Spalten) |
dplyr |
filter() |
wählt Beobachtungen (Zeilen) aus |
dplyr |
arrange() |
sortiert einen Datensatz nach einer bestimmten Variablen |
dplyr |
mutate() |
erstellt neue Variablen und ändert bereits vorhandene Variablen |
dplyr |
case_when() |
zum Rekodieren von vorhandenen Variablen |
forcats |
fct_recode() |
zum Rekodieren/Umbenennen von Faktorstufen |
dplyr |
group_by() |
ermöglicht Operationen an Teilmengen der Daten |
dplyr |
summarize() / summarise() |
fasst Daten zusammen |
Mit diesen Funktionen können sehr komplexe Datenmanipulationen durchgeführt werden und trotzdem bleibt der R Code relativ übersichtlich. Neben den oben genannten enthält dplyr noch viele weitere Funktionen. Dazu gehören z.B. Funktionen, die es ermöglichen, verschiedene Datensätze miteinander zu verbinden (zu “mergen”).
4.2 Der Pipe Operator
Wir haben schon festgestellt, dass Code schnell unübersichtlich werden kann, wenn wir eine Sequenz von Operationen ausführen. Dies führt zu verschachtelten Funktionsaufrufen.
Beispiel: Wir haben einen numerischen Vektor von \(n = 10\) Messwerten (hier zu Übungszwecken mit rnorm() aus normalverteilten Zufallszahlen generiert) und wollen diese zuerst zentrieren, dann die Standardabweichung berechnen, und anschliessend noch auf zwei Nachkommastellen runden.
set.seed(1283)
stichprobe <- rnorm(10, 24, 5)
stichprobe [1] 24.74984 21.91726 23.98551 19.63019 23.96428 22.83092 18.86240 19.08125
[9] 23.76589 21.88846Die gewünschte Berechnung der gerundeten Standardabweichung der zentrierten Werte können wir als verschachtelte Funktionsaufrufe durchführen:
round(sd(scale(stichprobe,
center = TRUE,
scale = FALSE)),
digits = 2)[1] 2.19Die Funktion scale(), sd() und round() werden nun der Reihe nach ausgeführt (von innen nach aussen), und zwar so, dass der Output einer Funktion der nächsten Funktion als Input übergeben wird.
Die Funktionen scale() und round() haben zusätzlich noch Argumente: center = TRUE, scale = FALSE, bzw. digits = 2. Dies ist zwar effizient, aber führt zu Code, der schwierig zu lesen ist.
Eine Alternative dazu wäre, die Zwischenschritte als Variablen zu speichern:
stichprobe_z <- scale(stichprobe, center = TRUE,
scale = FALSE)
sd_stichprobe_z <- sd(stichprobe_z)
sd_stichprobe_z_gerundet <- round(sd_stichprobe_z,
digits = 2)
sd_stichprobe_z_gerundet[1] 2.19So steht jeder der Teilschritte in einer eigenen Zeile und wir verstehen den Code ohne Probleme. Diese Methode erfordert jedoch, dass wir zwei Variablen definieren, die wir eigentlich gar nicht brauchen.
Es gibt nun aber eine sehr elegante Methode, um Funktionen nacheinander aufzurufen, ohne diese Funktionen ineinander verschachtelt schreiben zu müssen: wir benützen dafür den pipe Operator. Er sieht so aus:
|>und ist als Infix-Operator definiert. Das bedeutet, dass er zwischen zwei Objekten steht, ähnlich wie ein mathematischer Operator. Der Name pipe ist so zu verstehen, dass wir ein Objekt an eine Funktionen “weiterleiten” oder “übergeben”.
Dieser pipe Operator wird so oft verwendet, dass er schon eine eigene Tastenkombination hat: Cmd+Shift+M (MacOS) oder Ctrl+Shift+M (Windows und Linux).
Unser Beispiel von oben:
round(sd(scale(stichprobe,
center = TRUE,
scale = FALSE)),
digits = 2)[1] 2.19wird mit dem |> Operator zu:
stichprobe |>
scale(center = TRUE, scale = FALSE) |>
sd() |>
round(digits = 2)[1] 2.19Dieser Code ist so zu lesen:
- Wir beginnen mit dem Objekt
stichprobeund übergeben es mit|>als Argument an die Funktionscale() - Wir wenden
scale(), mit den zusätzlichen Argumentencenter = TRUE, scale = FALSEdarauf an, und übergeben den Output als Argument an die Funktionsd() - Wir wenden
sd()an (ohne weitere Argumente) und reichen den Output als Argument weiter anround() round(), mit dem weiteren Argumentdigits = 2, wird ausgeführt. Da kein weitererpipefolgt, wird der Output in die Konsole geschrieben.
Somit ist klar: wenn wir das Resultat weiterverwenden möchten, müssen wir es einer Variablen zuweisen:
sd_stichprobe_z_gerundet <- stichprobe |>
scale(center = TRUE, scale = FALSE) |>
sd() |>
round(digits = 2)
sd_stichprobe_z_gerundet[1] 2.19Wir übergeben also mit |> ein Objekt an eine Funktion. Wenn wir nichts weiter spezifizieren, ist dieses Objekt das erste Argument der Funktion. Die grossen Vorteile sind:
- Unser Code ist lesbarer.
- Wir mussten keine unnötigen Variablen definieren.
Syntax des Pipe Operators
Der |> Operator wird im Allgemeinen wie folgt verwendet. Nehmen wir an f(), g() und h() seien Funktionen, dann gilt:
x |> f()
# ist äquivalent zu
f(x)Wenn y ein weiteres Argument von f() ist, dann gilt:
x |> f(y)
# ist äquivalent zu
f(x, y)Wenn wir der Reihe nach f(), g() und h() anwenden, dann gilt:
x |> f() |> g() |> h()
# oder
x |>
f() |>
g() |>
h()
# ist äquivalent zu
h(g(f(x)))Wir müssen das Objekt x nicht als erstes Argument weitergeben, sondern können es an einer beliebigen Stelle der Funktion verwenden, an die x weitergegeben wird. Dafür brauchen wir den Argument-Platzhalter _. Dabei muss der Name des Arguments, bei dem der Platzhalter eingesetzt wird, immer explizit angegeben werden:
x |> f(y, argument = _)
# ist äquivalent zu
f(y, argument = x)Zum Beispiel könnten wir den oben in Kapitel 3.2 eingelesenen Datensatz zufriedenheit als letztes Argument data an die Funktion aggregate() pipen, um die Mittelwerte (Argument: FUN = mean) des Zufriedenheits-Ratings für die beiden Messzeitpunkten zu berechnen:
load(file = "data/zufriedenheit.Rda")
zufriedenheit# A tibble: 8 × 3
vpn messzeitpunkt zufriedenheit
<fct> <fct> <dbl>
1 vp_1 t1 64.3
2 vp_2 t1 65.1
3 vp_3 t1 54.2
4 vp_4 t1 54.5
5 vp_1 t2 67.2
6 vp_2 t2 75.6
7 vp_3 t2 84.6
8 vp_4 t2 73.9zufriedenheit |>
aggregate(zufriedenheit ~ messzeitpunkt, FUN = mean, data = _) messzeitpunkt zufriedenheit
1 t1 59.5325
2 t2 75.3425In den meisten Fällen ist jedoch das Objekt, welches übergeben wird, gleichzeitig auch das erste Argument der nächsten Funktion (vor allem für die tidyverse Funktionen), so dass wir diesen Platzhalter selten brauchen.
Wenn wir mit den Funktionen der tidyr und dplyr Packages arbeiten, werden wir diesen Operator sehr häufig benutzen. Ein weiterer Grund, sich damit anzufreunden, ist, dass dieser immer häufiger Verwendung findet und sehr viele Beispiele im Internet (z.B. auf Stackoverflow) den |> Operator benutzen.
4.3 Reshaping: tidyr
Um einen Datensatz zu transformieren (reshaping) brauchen wir zwei Funktionen: pivot_longer() und pivot_wider(), beide befinden sich im Package tidyr.
library(tidyr)4.3.1 pivot_longer()
Wir benutzen pivot_longer(), wenn wir einen wide Datensatz zu einem long Datensatz konvertieren möchten. pivot_longer() wird also dazu verwendet, mehrere Spalten, die die Stufen eines Faktors repräsentieren, zu einer Spalte zusammenzufügen, welche den Faktor selbst repräsentiert. Die Werte in den ursprünglichen Variablen werden in einer Werte-Variable zusammengefasst.
Die Syntax von pivot_longer() sieht so aus:
pivot_longer(data, cols, names_to, values_to)oder mit |>
data |>
pivot_longer(cols, names_to, values_to)Die Argumente haben die folgende Bedeutung:
data: ein data frame
cols: die Spalten, deren Werte in einer Spalte zusammengeführt werden sollen
names_to: "Name" (string) der neuen Spalte, die aus den ausgewählten Spaltennamen
erstellt wird
values_to "Name" (string) der Spalte, die aus den Beobachtungen in den Zellen
erstellt wirdSehen wir uns noch einmal das oben verwendete Beispiel an. Wir haben einen wide Datensatz (mit Namen df) mit zwei Personen, die zu drei Zeitpunkten gemessen wurden.
df# A tibble: 2 × 4
name t1 t2 t3
<fct> <dbl> <dbl> <dbl>
1 Marshall 4 5 7
2 Skye 3 6 7Wir nehmen an, dass t1, t2 und t3 keine wirklich getrennten Variablen sind, sondern dass sie als Stufen eines Zeitfaktors betrachtet werden können (z.B. als drei Zeitpunkte in einem Messwiederholungsdesign). Die Werte in den Spalten t1, t2 und t3 beziehen sich auf die gleiche Art von Messung (z.B. Punktzahl) und sollten daher tatsächlich durch eine Variable in einem long Datensatz repräsentiert werden. Das sind also die im Argument cols zu bestimmenden Variablen/Spalten.
Wir möchten, dass unser neuer Faktor den Namen zeit erhält und die gemessene Variable punkte genannt wird.
Die name-Variable sollte ignoriert werden, d.h. sie sollte bei der Umstrukturierung nicht mit benutzt werden (also von der Funktion nicht als weiterer Messzeitpunkt betrachtet werden).
library(tidyr)
df_long <- df |>
pivot_longer(!name, names_to = "zeit", values_to = "punkte")df_long# A tibble: 6 × 3
name zeit punkte
<fct> <chr> <dbl>
1 Marshall t1 4
2 Marshall t2 5
3 Marshall t3 7
4 Skye t1 3
5 Skye t2 6
6 Skye t3 7zeit sollte im nächsten Schritt noch als Faktor definiert werden:
df_long$zeit <- as.factor(df_long$zeit)df_long# A tibble: 6 × 3
name zeit punkte
<fct> <fct> <dbl>
1 Marshall t1 4
2 Marshall t2 5
3 Marshall t3 7
4 Skye t1 3
5 Skye t2 6
6 Skye t3 7Eine weitere Eigenschaft von pivot_longer() ist, dass die Werte derjenigen Variablen, die nicht Teil des Reshapings sind (in unserem Fall also einzig die Variable name) wiederholt werden. Die Zeilen 1-3 beinhalten jetzt also Beobachtungen, die zu Marshall gehören und die Zeilen 4-6 Beobachtungen, die zu Skye gehören.
Der Aufruf der Funktion pivot_longer() hätte auch expliziter geschrieben werden können:
df |>
pivot_longer(cols = !name,
names_to = "zeit",
values_to = "punkte")Im cols-Argument haben wir !name verwendet: so werden alle Spalten ausser name für die wide-to-long-Transformation verwendet. Wir hätten stattdessen auch direkt die drei Zeit-Spalten auswählen können:
df |>
pivot_longer(cols = c(t1, t2, t3),
names_to = "zeit",
values_to = "punkte")# A tibble: 6 × 3
name zeit punkte
<fct> <chr> <dbl>
1 Marshall t1 4
2 Marshall t2 5
3 Marshall t3 7
4 Skye t1 3
5 Skye t2 6
6 Skye t3 7Wir werden weitere und allgemeinere Möglichkeiten für die Auswahl von Spalten/Variablen eines Datensatzes kennenlernen, wenn wir uns das dplyr Package ansehen.
Beispiel
Schauen wir uns ein weiteres Beispiel an, diesmal unter Verwendung des therapie-Datensatzes, den wir zunächst von Github herunterladen und anschliessend in unserem data-Ordner abspeichern: therapie.sav.
Jetzt lesen wir den Datensatz lokal aus unserem data-Ordner ein und konvertieren die dort vorhandenen Faktoren:
library(haven)
therapie <- read_sav("data/therapie.sav")
therapie$id <- as_factor(therapie$id)
therapie$gruppe <- as_factor(therapie$gruppe)therapie# A tibble: 100 × 4
id gruppe pretest posttest
<fct> <fct> <dbl> <dbl>
1 1 Kontrollgruppe 4.29 3.21
2 2 Kontrollgruppe 6.18 5.99
3 3 Kontrollgruppe 3.93 4.17
4 4 Kontrollgruppe 5.06 4.76
5 5 Kontrollgruppe 6.45 5.64
6 6 Kontrollgruppe 4.49 4.67
7 7 Kontrollgruppe 4.60 4.24
8 8 Kontrollgruppe 4.46 3.34
9 9 Kontrollgruppe 4.76 4.11
10 10 Kontrollgruppe 5.12 5.29
# ℹ 90 more rowsDie Struktur dieses Datensatzes ähnelt der des obigen Beispiels. Wir wollen die Spalten pretest und posttest kombinieren, da sie Stufen eines gemeinsamen Faktors zeit sind. Die Daten in den Zellen repräsentieren also wiederholte Messungen. Da die Variablen ausser den Messzeitpunkten pretest und posttest keine weiteren Informationen aufweisen, können wir aus dem Datensatz allein nicht erkennen, was eigentlich die gemessene (psychologische) Variable ist. Im obigen Einführungsbeispiel haben wir die beim Pivotieren gebildete Variable einfach punkte genannt. Jetzt wollen wir der Variable aber einen Namen geben, der dem Inhalt der Messungen entspricht. Dafür benötigen wir die Zusatzinformation, dass es sich bei den Werten unter pretest und posttest um eine Skala psychischer Stresssymptome handelt, die auf einer Skala von 0 bis 7 gemessen wurden. Wir wollen die Variable daher symptome nennen.
Ausserdem sollen die Variablen id und gruppe für das Pivotieren ignoriert werden, aber im Datensatz verbleiben.
Führen wir die wide-to-long-Transformation durch:
therapie_long <- therapie |>
pivot_longer(c("pretest", "posttest"),
names_to = "zeit",
values_to = "symptome")
# zeit sollte ein Faktor sein
therapie_long$zeit <- factor(therapie_long$zeit,
levels = c("pretest", "posttest"))
# Hier definieren wir explizit 'pretest' als erste und 'posttest' als zweite
# Faktorstufe, weil es inhaltlich und für spätere Datenanalysen Sinn macht, den
# zeitlich früher gelegenen Messzeitpunkt als erste Faktorstufe zu bestimmen.
# Ohne explizite Definition über das levels-Argument wäre hier 'posttest' die
# erste Faktorstufe (weil alphabetisch vor 'pretest').therapie_long# A tibble: 200 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe pretest 4.29
2 1 Kontrollgruppe posttest 3.21
3 2 Kontrollgruppe pretest 6.18
4 2 Kontrollgruppe posttest 5.99
5 3 Kontrollgruppe pretest 3.93
6 3 Kontrollgruppe posttest 4.17
7 4 Kontrollgruppe pretest 5.06
8 4 Kontrollgruppe posttest 4.76
9 5 Kontrollgruppe pretest 6.45
10 5 Kontrollgruppe posttest 5.64
# ℹ 190 more rows4.3.2 pivot_wider()
pivot_wider() ist das Gegenteil von pivot_longer(). Diese Funktion nimmt einen Faktor und eine Messvariable und “verteilt” die Werte der Messvariable über neue Spalten, welche die Stufen des Faktors repräsentieren. Dies bedeutet, dass wir pivot_wider() verwenden, wenn wir aus einem long Datensatz einen wide Datensatz machen wollen.
Die pivot_wider() Syntax sieht so aus:
pivot_wider(data, names_from, values_from)
# oder
data |>
pivot_wider(names_from, values_from)An unserem einfachen Beispiel illustriert bedeutet dies, dass wir neue Variablen für jede Stufe des Faktors zeit erstellen wollen, uns zwar unter Verwendung der Werte der Variable punkte.
df_long# A tibble: 6 × 3
name zeit punkte
<fct> <fct> <dbl>
1 Marshall t1 4
2 Marshall t2 5
3 Marshall t3 7
4 Skye t1 3
5 Skye t2 6
6 Skye t3 7df_wide <- df_long |>
pivot_wider(names_from = zeit, values_from = punkte)df_wide# A tibble: 2 × 4
name t1 t2 t3
<fct> <dbl> <dbl> <dbl>
1 Marshall 4 5 7
2 Skye 3 6 7df_wide sieht genau so aus wie der ursprüngliche Datensatz df. Wir können noch prüfen, ob beide wirklich äquivalent sind:
all.equal(df, df_wide) [1] TRUEBeispiel
Nun konvertieren wir den therapie-Datensatz von long zurück zu wide:
therapie_long# A tibble: 200 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe pretest 4.29
2 1 Kontrollgruppe posttest 3.21
3 2 Kontrollgruppe pretest 6.18
4 2 Kontrollgruppe posttest 5.99
5 3 Kontrollgruppe pretest 3.93
6 3 Kontrollgruppe posttest 4.17
7 4 Kontrollgruppe pretest 5.06
8 4 Kontrollgruppe posttest 4.76
9 5 Kontrollgruppe pretest 6.45
10 5 Kontrollgruppe posttest 5.64
# ℹ 190 more rowstherapie_wide <- therapie_long |>
pivot_wider(names_from = zeit, values_from = symptome)therapie_wide# A tibble: 100 × 4
id gruppe pretest posttest
<fct> <fct> <dbl> <dbl>
1 1 Kontrollgruppe 4.29 3.21
2 2 Kontrollgruppe 6.18 5.99
3 3 Kontrollgruppe 3.93 4.17
4 4 Kontrollgruppe 5.06 4.76
5 5 Kontrollgruppe 6.45 5.64
6 6 Kontrollgruppe 4.49 4.67
7 7 Kontrollgruppe 4.60 4.24
8 8 Kontrollgruppe 4.46 3.34
9 9 Kontrollgruppe 4.76 4.11
10 10 Kontrollgruppe 5.12 5.29
# ℹ 90 more rows4.3.3 Fehlende Werte auschliessen: drop_na()
Eine ganz wichtige Funktion im tidyr Package ist drop_na(). Damit können wir alle Zeilen löschen, welche fehlende Werte haben.
Als Illustration dient dieses Beispiel:
df_1 <- tibble(var1 = factor(c("a", NA, "b", "b")), var2 = c(1, NA, 2, 3), var3 = c(NA, 21, 24, NA))
df_1# A tibble: 4 × 3
var1 var2 var3
<fct> <dbl> <dbl>
1 a 1 NA
2 <NA> NA 21
3 b 2 24
4 b 3 NAdf_1 |>
drop_na() # A tibble: 1 × 3
var1 var2 var3
<fct> <dbl> <dbl>
1 b 2 24Wir haben einen Faktor (var1) und zwei numerische Variablen (var2 und var3). Für viele statistische Analysen benötigen wir vollständige Datensätze, d.h. Datensätze ohne fehlende Werte/NAs. In diesem Fall bleibt von den vier Beobachtungen nur noch eine übrig, da nur Zeile 3 des Datensatzes kein einziges NA aufweist.
Angenommen, die für eine bestimmte Analyse interessierenden Variablen sind var1 und var2, während var3 (könnte z.B. die Altersvariable sein), keine Rolle spielt.
Dann wäre es sinnvoll, zunächst einen Subdatensatz zu bilden, der nur die ersten beiden Variablen enthält, und erst dann drop_na() auszuführen:
df_1a <- df_1[1:2]
df_1a |>
drop_na() # A tibble: 3 × 2
var1 var2
<fct> <dbl>
1 a 1
2 b 2
3 b 3So verbleiben drei Beobachtungen für die nachfolgende Datenanalyse (alle ausser die zweite Zeile/Person aus df_1, die sowohl in var1 als auch in var2 ein NA aufweist).
Diese Funktion ist sehr nützlich, sollte aber wie im Beispiel gezeigt vorsichtig verwendet werden, denn sonst löscht man ggf. Zeilen, die NAs in Variablen besitzen, die für die vorliegende Fragestellung nicht relevant sind.
4.4 Data Wrangling: dplyr
Nun sind wir in der Lage, Datensätze von wide zu long und umgekehrt zu transformieren, aber damit ist unsere Arbeit noch nicht getan. Die meisten Datensätze müssen bearbeitet werden, bevor sie analysiert werden können: Wir müssen z.B. Fälle und/oder Variablen auswählen, Werte recodieren, Variablen umbenennen, nach bestimmten Variablen sortieren, neue Variablen bilden, Datensätze nach Gruppierungsvariablen aufteilen oder Variablen zusammenfassen. Im Englischen wird diese Art von Arbeit an und mit Daten oft als data wrangling bezeichnet.
Das dplyr Package stellt Funktionen für alle diese Aufgaben zur Verfügung (und noch viele mehr, wir betrachten hier nur eine Auswahl). dplyr besteht sozusagen aus Verben (Funktionen) für all diese Operationen, und diese Funktionen können - je nach Bedarf - auf sehr elegante Weise zusammengesetzt werden.
Für den Rest dieses Kapitels arbeiten wir mit long Datensätzen. Falls ein Datensatz wide ist, sollte er zu long konvertiert werden.
Wir laden zuerst das dplyr Package:
library(dplyr)Wir sehen uns nun der Reihe nach die verschiedenen Funktionen und deren Verwendung an. Wir verwenden immer den |> Operator. Der Input Dataframe ist dabei immer als erstes Argument der Funktion zu verstehen. Für die Beispiele verwenden wir die Datensätze df_long.Rda, therapie_long.Rda, therapie_wide.Rda sowie alkohol_aggression.csv und beispieldaten.sav. Speichern Sie die heruntergeladenen Datenfiles in Ihrem data-Ordner ab.
Während die ersten drei bereits weiter oben geladen und verwendet wurden, lesen wir die letzten beiden beim jeweiligen Beispiel ein.
Bei allen unten stehenden Beispielen gilt: wenn wir das Resultat weiterverwenden möchten, müssen wir den Output einer neuen Variablen zuweisen.
4.4.1 Variablen umbennen mit rename()
Variablen können mit rename() umbenannt werden. Die nicht umbenannten Variablen verbleiben im Datensatz.
Syntax:
df |> rename(neuer_name = alter_name)Beispiel
# "zeit" umbennen in "messzeitpunkt"
therapie_long |>
rename(messzeitpunkt = zeit)# A tibble: 200 × 4
id gruppe messzeitpunkt symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe pretest 4.29
2 1 Kontrollgruppe posttest 3.21
3 2 Kontrollgruppe pretest 6.18
4 2 Kontrollgruppe posttest 5.99
5 3 Kontrollgruppe pretest 3.93
6 3 Kontrollgruppe posttest 4.17
7 4 Kontrollgruppe pretest 5.06
8 4 Kontrollgruppe posttest 4.76
9 5 Kontrollgruppe pretest 6.45
10 5 Kontrollgruppe posttest 5.64
# ℹ 190 more rows4.4.2 Variablen auswählen mit select()
Mit der Funktion select() wählen wir Variablen aus einem Datensatz aus.
Syntax:
# df steht für data frame
select(df, variable1, variable2, !variable3)
# oder
df |> select(variable1, variable2, !variable3)select() wählt hier aus dem Dataframe df die Variablen variable1 und variable2 aus, variable3 wird weggelassen.
Beispiele
# nur ID
df_long |> select(name)# A tibble: 6 × 1
name
<fct>
1 Marshall
2 Marshall
3 Marshall
4 Skye
5 Skye
6 Skye # Messzeitpunkt und Score
df_long |> select(zeit, punkte)# A tibble: 6 × 2
zeit punkte
<fct> <dbl>
1 t1 4
2 t2 5
3 t3 7
4 t1 3
5 t2 6
6 t3 7# oder
df_long |> select(!name)# A tibble: 6 × 2
zeit punkte
<fct> <dbl>
1 t1 4
2 t2 5
3 t3 7
4 t1 3
5 t2 6
6 t3 7therapie_long |>
select(id, gruppe, symptome)# A tibble: 200 × 3
id gruppe symptome
<fct> <fct> <dbl>
1 1 Kontrollgruppe 4.29
2 1 Kontrollgruppe 3.21
3 2 Kontrollgruppe 6.18
4 2 Kontrollgruppe 5.99
5 3 Kontrollgruppe 3.93
6 3 Kontrollgruppe 4.17
7 4 Kontrollgruppe 5.06
8 4 Kontrollgruppe 4.76
9 5 Kontrollgruppe 6.45
10 5 Kontrollgruppe 5.64
# ℹ 190 more rows# wählt alle Variablen von gruppe bis symptome aus
# (also auch die Variablen dazwischen)
therapie_long |>
select(gruppe:symptome)# A tibble: 200 × 3
gruppe zeit symptome
<fct> <fct> <dbl>
1 Kontrollgruppe pretest 4.29
2 Kontrollgruppe posttest 3.21
3 Kontrollgruppe pretest 6.18
4 Kontrollgruppe posttest 5.99
5 Kontrollgruppe pretest 3.93
6 Kontrollgruppe posttest 4.17
7 Kontrollgruppe pretest 5.06
8 Kontrollgruppe posttest 4.76
9 Kontrollgruppe pretest 6.45
10 Kontrollgruppe posttest 5.64
# ℹ 190 more rows# Für die gleichzeitige Auswahl und Umbenennung
# einer Variable verwendet man =. Der neue Name
# steht auf der linken Seite des =, die alte
# Variable auf der rechten Seite:
therapie_long |>
select(messzeitpunkt = zeit)# A tibble: 200 × 1
messzeitpunkt
<fct>
1 pretest
2 posttest
3 pretest
4 posttest
5 pretest
6 posttest
7 pretest
8 posttest
9 pretest
10 posttest
# ℹ 190 more rowsHilfsfunktionen für select()
Für die Auswahl von Variablen gibt es eine Reihe von sogenannten “helper functions”:
# einschliessen
therapie_long |> select(starts_with("gr"))# A tibble: 200 × 1
gruppe
<fct>
1 Kontrollgruppe
2 Kontrollgruppe
3 Kontrollgruppe
4 Kontrollgruppe
5 Kontrollgruppe
6 Kontrollgruppe
7 Kontrollgruppe
8 Kontrollgruppe
9 Kontrollgruppe
10 Kontrollgruppe
# ℹ 190 more rowstherapie_long |> select(ends_with("me"))# A tibble: 200 × 1
symptome
<dbl>
1 4.29
2 3.21
3 6.18
4 5.99
5 3.93
6 4.17
7 5.06
8 4.76
9 6.45
10 5.64
# ℹ 190 more rowstherapie_long |> select(contains("p"))# A tibble: 200 × 2
gruppe symptome
<fct> <dbl>
1 Kontrollgruppe 4.29
2 Kontrollgruppe 3.21
3 Kontrollgruppe 6.18
4 Kontrollgruppe 5.99
5 Kontrollgruppe 3.93
6 Kontrollgruppe 4.17
7 Kontrollgruppe 5.06
8 Kontrollgruppe 4.76
9 Kontrollgruppe 6.45
10 Kontrollgruppe 5.64
# ℹ 190 more rowsvars <- c("gruppe", "symptome")
# einschliessendes ODER mit one_of()
therapie_long |> select(one_of(vars))# A tibble: 200 × 2
gruppe symptome
<fct> <dbl>
1 Kontrollgruppe 4.29
2 Kontrollgruppe 3.21
3 Kontrollgruppe 6.18
4 Kontrollgruppe 5.99
5 Kontrollgruppe 3.93
6 Kontrollgruppe 4.17
7 Kontrollgruppe 5.06
8 Kontrollgruppe 4.76
9 Kontrollgruppe 6.45
10 Kontrollgruppe 5.64
# ℹ 190 more rows# ausschliessen
therapie_long |> select(!starts_with("gr"))# A tibble: 200 × 3
id zeit symptome
<fct> <fct> <dbl>
1 1 pretest 4.29
2 1 posttest 3.21
3 2 pretest 6.18
4 2 posttest 5.99
5 3 pretest 3.93
6 3 posttest 4.17
7 4 pretest 5.06
8 4 posttest 4.76
9 5 pretest 6.45
10 5 posttest 5.64
# ℹ 190 more rowstherapie_long |> select(!ends_with("me"))# A tibble: 200 × 3
id gruppe zeit
<fct> <fct> <fct>
1 1 Kontrollgruppe pretest
2 1 Kontrollgruppe posttest
3 2 Kontrollgruppe pretest
4 2 Kontrollgruppe posttest
5 3 Kontrollgruppe pretest
6 3 Kontrollgruppe posttest
7 4 Kontrollgruppe pretest
8 4 Kontrollgruppe posttest
9 5 Kontrollgruppe pretest
10 5 Kontrollgruppe posttest
# ℹ 190 more rowstherapie_long |> select(!contains("p"))# A tibble: 200 × 2
id zeit
<fct> <fct>
1 1 pretest
2 1 posttest
3 2 pretest
4 2 posttest
5 3 pretest
6 3 posttest
7 4 pretest
8 4 posttest
9 5 pretest
10 5 posttest
# ℹ 190 more rowsvars <- c("gruppe", "symptome")
therapie_long |> select(!one_of(vars))# A tibble: 200 × 2
id zeit
<fct> <fct>
1 1 pretest
2 1 posttest
3 2 pretest
4 2 posttest
5 3 pretest
6 3 posttest
7 4 pretest
8 4 posttest
9 5 pretest
10 5 posttest
# ℹ 190 more rows# Datensatz 'beispieldaten.sav' importieren und alle Items von swk1 bis swk12 auswählen
beispieldaten <- read_sav("data/beispieldaten.sav")
beispieldaten |>
select(num_range("swk", 1:12))# A tibble: 286 × 12
swk1 swk2 swk3 swk4 swk5 swk6 swk7 swk8 swk9 swk10 swk11 swk12
<dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 2 5 6 4 7 5 5 6 6 4 4 5
2 4 6 4 4 6 5 5 6 5 4 4 4
3 5 4 5 6 5 7 7 6 4 5 3 3
4 3 6 6 5 7 7 6 6 7 7 3 4
5 5 6 6 5 7 6 5 5 7 6 4 4
6 3 5 5 4 5 7 5 6 6 5 3 5
7 4 3 5 4 6 7 1 6 5 7 3 6
8 5 4 7 7 7 7 NA 7 7 6 4 3
9 6 6 5 5 7 6 5 6 6 6 6 6
10 5 3 4 5 5 5 5 4 5 5 5 6
# ℹ 276 more rowsDamit können Variablen anhand von Suchkriterien ausgewählt oder ausgeschlossen werden. Wir werden in späteren Kapiteln weitere Beispiele dafür sehen.
# alle numerischen Variablen auswählen
therapie_wide |>
select(where(is.numeric))# A tibble: 100 × 2
pretest posttest
<dbl> <dbl>
1 4.29 3.21
2 6.18 5.99
3 3.93 4.17
4 5.06 4.76
5 6.45 5.64
6 4.49 4.67
7 4.60 4.24
8 4.46 3.34
9 4.76 4.11
10 5.12 5.29
# ℹ 90 more rows4.4.3 Reihenfolge der Variablen verändern mit relocate()
Mit der Funktion relocate() verändern wir die Reihenfolge der Variablen. Per default werden die Variablen an den Anfang des Datensatzes verschoben. Mit den Argumenten .after und .before kann spezifiziert werden, wohin die Variablen verschoben werden sollen.
Beispiele
# pretest und posttest an den Anfang verschieben
therapie_wide |>
relocate(pretest, posttest)# A tibble: 100 × 4
pretest posttest id gruppe
<dbl> <dbl> <fct> <fct>
1 4.29 3.21 1 Kontrollgruppe
2 6.18 5.99 2 Kontrollgruppe
3 3.93 4.17 3 Kontrollgruppe
4 5.06 4.76 4 Kontrollgruppe
5 6.45 5.64 5 Kontrollgruppe
6 4.49 4.67 6 Kontrollgruppe
7 4.60 4.24 7 Kontrollgruppe
8 4.46 3.34 8 Kontrollgruppe
9 4.76 4.11 9 Kontrollgruppe
10 5.12 5.29 10 Kontrollgruppe
# ℹ 90 more rows# gruppe vor posttest verschieben
therapie_wide |>
relocate(gruppe, .before = posttest)# A tibble: 100 × 4
id pretest gruppe posttest
<fct> <dbl> <fct> <dbl>
1 1 4.29 Kontrollgruppe 3.21
2 2 6.18 Kontrollgruppe 5.99
3 3 3.93 Kontrollgruppe 4.17
4 4 5.06 Kontrollgruppe 4.76
5 5 6.45 Kontrollgruppe 5.64
6 6 4.49 Kontrollgruppe 4.67
7 7 4.60 Kontrollgruppe 4.24
8 8 4.46 Kontrollgruppe 3.34
9 9 4.76 Kontrollgruppe 4.11
10 10 5.12 Kontrollgruppe 5.29
# ℹ 90 more rows# id nach gruppe verschieben
therapie_wide |>
relocate(id, .after = gruppe)# A tibble: 100 × 4
gruppe id pretest posttest
<fct> <fct> <dbl> <dbl>
1 Kontrollgruppe 1 4.29 3.21
2 Kontrollgruppe 2 6.18 5.99
3 Kontrollgruppe 3 3.93 4.17
4 Kontrollgruppe 4 5.06 4.76
5 Kontrollgruppe 5 6.45 5.64
6 Kontrollgruppe 6 4.49 4.67
7 Kontrollgruppe 7 4.60 4.24
8 Kontrollgruppe 8 4.46 3.34
9 Kontrollgruppe 9 4.76 4.11
10 Kontrollgruppe 10 5.12 5.29
# ℹ 90 more rows4.4.4 Beobachtungen (Fälle) auswählen mit filter()
Beobachtungen oder Fälle (Zeilen) werden mit filter() ausgewählt, d.h. wir können damit Fälle auswählen, welche gewisse Bedingungen erfüllen.
Es können auch mehrere Bedingungen mit logischen Operatoren verknüpft werden:
Syntax:
df |> filter(variable1 < WERT1 & variable2 == WERT2)Beispiele
# nur Kontrollgruppe auswählen
therapie_long |>
filter(gruppe == "Kontrollgruppe")# A tibble: 100 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe pretest 4.29
2 1 Kontrollgruppe posttest 3.21
3 2 Kontrollgruppe pretest 6.18
4 2 Kontrollgruppe posttest 5.99
5 3 Kontrollgruppe pretest 3.93
6 3 Kontrollgruppe posttest 4.17
7 4 Kontrollgruppe pretest 5.06
8 4 Kontrollgruppe posttest 4.76
9 5 Kontrollgruppe pretest 6.45
10 5 Kontrollgruppe posttest 5.64
# ℹ 90 more rows# nur posttest
therapie_long |>
filter(zeit == "posttest")# A tibble: 100 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe posttest 3.21
2 2 Kontrollgruppe posttest 5.99
3 3 Kontrollgruppe posttest 4.17
4 4 Kontrollgruppe posttest 4.76
5 5 Kontrollgruppe posttest 5.64
6 6 Kontrollgruppe posttest 4.67
7 7 Kontrollgruppe posttest 4.24
8 8 Kontrollgruppe posttest 3.34
9 9 Kontrollgruppe posttest 4.11
10 10 Kontrollgruppe posttest 5.29
# ℹ 90 more rows# nur pretest
therapie_long |>
filter(zeit != "posttest")# A tibble: 100 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe pretest 4.29
2 2 Kontrollgruppe pretest 6.18
3 3 Kontrollgruppe pretest 3.93
4 4 Kontrollgruppe pretest 5.06
5 5 Kontrollgruppe pretest 6.45
6 6 Kontrollgruppe pretest 4.49
7 7 Kontrollgruppe pretest 4.60
8 8 Kontrollgruppe pretest 4.46
9 9 Kontrollgruppe pretest 4.76
10 10 Kontrollgruppe pretest 5.12
# ℹ 90 more rows# nur symptome >= 5
therapie_long |>
filter(symptome >= 5)# A tibble: 66 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 2 Kontrollgruppe pretest 6.18
2 2 Kontrollgruppe posttest 5.99
3 4 Kontrollgruppe pretest 5.06
4 5 Kontrollgruppe pretest 6.45
5 5 Kontrollgruppe posttest 5.64
6 10 Kontrollgruppe pretest 5.12
7 10 Kontrollgruppe posttest 5.29
8 11 Kontrollgruppe pretest 6.04
9 13 Kontrollgruppe posttest 5.16
10 19 Kontrollgruppe pretest 5.20
# ℹ 56 more rows# nur symptome mit Werten zwischen 3 und 5
therapie_long |>
filter(symptome >= 3 & symptome <= 5)# A tibble: 130 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe pretest 4.29
2 1 Kontrollgruppe posttest 3.21
3 3 Kontrollgruppe pretest 3.93
4 3 Kontrollgruppe posttest 4.17
5 4 Kontrollgruppe posttest 4.76
6 6 Kontrollgruppe pretest 4.49
7 6 Kontrollgruppe posttest 4.67
8 7 Kontrollgruppe pretest 4.60
9 7 Kontrollgruppe posttest 4.24
10 8 Kontrollgruppe pretest 4.46
# ℹ 120 more rows# nur Personen mit id 3 und 5
therapie_long |>
filter(id == 3 | id == 5)# A tibble: 4 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 3 Kontrollgruppe pretest 3.93
2 3 Kontrollgruppe posttest 4.17
3 5 Kontrollgruppe pretest 6.45
4 5 Kontrollgruppe posttest 5.64# Alternative dazu (mit dem %in% Operator)
therapie_long |>
filter(id %in% c(3, 5))# A tibble: 4 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 3 Kontrollgruppe pretest 3.93
2 3 Kontrollgruppe posttest 4.17
3 5 Kontrollgruppe pretest 6.45
4 5 Kontrollgruppe posttest 5.644.4.5 Beobachtungen (Fälle) sortieren mit arrange()
Mit der arrange() Funktion können wir Beobachtungen sortieren, entweder in aufsteigender oder in absteigender Reihenfolge.
# aufsteigend
therapie_long |>
arrange(id)# A tibble: 200 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 Kontrollgruppe pretest 4.29
2 1 Kontrollgruppe posttest 3.21
3 2 Kontrollgruppe pretest 6.18
4 2 Kontrollgruppe posttest 5.99
5 3 Kontrollgruppe pretest 3.93
6 3 Kontrollgruppe posttest 4.17
7 4 Kontrollgruppe pretest 5.06
8 4 Kontrollgruppe posttest 4.76
9 5 Kontrollgruppe pretest 6.45
10 5 Kontrollgruppe posttest 5.64
# ℹ 190 more rows# absteigend
therapie_long |>
arrange(desc(id))# A tibble: 200 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 100 Therapiegruppe pretest 4.77
2 100 Therapiegruppe posttest 4.50
3 99 Therapiegruppe pretest 4.66
4 99 Therapiegruppe posttest 3.80
5 98 Therapiegruppe pretest 4.36
6 98 Therapiegruppe posttest 3.91
7 97 Therapiegruppe pretest 4.53
8 97 Therapiegruppe posttest 4.36
9 96 Therapiegruppe pretest 5.15
10 96 Therapiegruppe posttest 5.05
# ℹ 190 more rows4.4.6 Neue Variablen erstellen mit mutate()
Neue Variablen können mit mutate() aus schon bestehenden Variablen gebildet werden.
Syntax:
df |> mutate(neue_variable_1 = FORMEL_1,
neue_variable_2 = FORMEL_2)Beispiele
In Fragebogenstudien kommt es häufig vor, dass man für jede Person aus mehreren Variablen/Items einen Mittelwert berechnen will, z.B. um einige der Selbstwirksamkeitsitems aus dem Beispieldatensatz zu einer Skala zusammenzufassen:
# Neue Variable mit dem Mittelwert der Items swk4 bis swk7 erstellen
beispieldaten |>
select(num_range("swk", 4:7)) |>
mutate(swk_mean = (swk4 + swk5 + swk6 + swk7) / 4)# A tibble: 286 × 5
swk4 swk5 swk6 swk7 swk_mean
<dbl> <dbl> <dbl> <dbl> <dbl>
1 4 7 5 5 5.25
2 4 6 5 5 5
3 6 5 7 7 6.25
4 5 7 7 6 6.25
5 5 7 6 5 5.75
6 4 5 7 5 5.25
7 4 6 7 1 4.5
8 7 7 7 NA NA
9 5 7 6 5 5.75
10 5 5 5 5 5
# ℹ 276 more rowsAn diesem Beispiel wird ersichtlich, dass die Berechnung des Mittelwerts über eine Formel nicht funktioniert, wenn NAs (fehlende Werte) vorhanden sind. Da in Zeile 8 kein Wert für swk7 vorhanden ist, kann in dieser Zeile kein Mittelwert berechnet werden.
Die Verwendung von mean() mit dem Argument na.rm = TRUE ist auch nicht möglich, da mean() dann den Mittelwert über alle Personen UND alle Variablen berechnet (und nicht pro Person den Mittelwert über die Variablen).
beispieldaten |>
select(num_range("swk", 4:7)) |>
mutate(swk_mean = mean(c(swk4, swk5, swk6, swk7), na.rm = TRUE))# A tibble: 286 × 5
swk4 swk5 swk6 swk7 swk_mean
<dbl> <dbl> <dbl> <dbl> <dbl>
1 4 7 5 5 5.58
2 4 6 5 5 5.58
3 6 5 7 7 5.58
4 5 7 7 6 5.58
5 5 7 6 5 5.58
6 4 5 7 5 5.58
7 4 6 7 1 5.58
8 7 7 7 NA 5.58
9 5 7 6 5 5.58
10 5 5 5 5 5.58
# ℹ 276 more rowsDie Funktion rowwise() aus dem dplyr-Package kann uns hier weiterhelfen.
beispieldaten |>
select(num_range("swk", 4:7)) |>
rowwise() |>
mutate(swk_mean = mean(c(swk4, swk5, swk6, swk7), na.rm = TRUE))# A tibble: 286 × 5
# Rowwise:
swk4 swk5 swk6 swk7 swk_mean
<dbl> <dbl> <dbl> <dbl> <dbl>
1 4 7 5 5 5.25
2 4 6 5 5 5
3 6 5 7 7 6.25
4 5 7 7 6 6.25
5 5 7 6 5 5.75
6 4 5 7 5 5.25
7 4 6 7 1 4.5
8 7 7 7 NA 7
9 5 7 6 5 5.75
10 5 5 5 5 5
# ℹ 276 more rowsDie Verwendung von rowwise() führt dazu, dass mean(na.rm = TRUE) den Mittelwert für jede Zeile berechnet. In Zeile 8 wurde nun also der Mittelwert über die verbliebenen 3 Items berechnet.
Mit mutate() können wir auch eine bestehende Variable umberechnen. In folgendem Beispiel rechnen wir die Symptom-Werte (Skala von 0 bis 7) in Prozentwerte um.
therapie_long |>
mutate(symptome_p = round(symptome / 7 * 100, digits = 1))# A tibble: 200 × 5
id gruppe zeit symptome symptome_p
<fct> <fct> <fct> <dbl> <dbl>
1 1 Kontrollgruppe pretest 4.29 61.2
2 1 Kontrollgruppe posttest 3.21 45.8
3 2 Kontrollgruppe pretest 6.18 88.2
4 2 Kontrollgruppe posttest 5.99 85.5
5 3 Kontrollgruppe pretest 3.93 56.2
6 3 Kontrollgruppe posttest 4.17 59.6
7 4 Kontrollgruppe pretest 5.06 72.3
8 4 Kontrollgruppe posttest 4.76 68.1
9 5 Kontrollgruppe pretest 6.45 92.2
10 5 Kontrollgruppe posttest 5.64 80.6
# ℹ 190 more rowsMit dem Argument .keep = c("all", "used", "unused", "none") kann kontrolliert werden, welche Variablen im Output erscheinen. .keep = "used" führt z.B. dazu, dass neben der neu gebildeten Variable nur diejenigen Variablen, die für die Erstellung der neuen Variable verwendet wurden, im Output erscheinen.
therapie_long |>
mutate(symptome_p = round(symptome / 7 * 100, digits = 1),
.keep = "used")# A tibble: 200 × 2
symptome symptome_p
<dbl> <dbl>
1 4.29 61.2
2 3.21 45.8
3 6.18 88.2
4 5.99 85.5
5 3.93 56.2
6 4.17 59.6
7 5.06 72.3
8 4.76 68.1
9 6.45 92.2
10 5.64 80.6
# ℹ 190 more rows.keep = "none" führt dazu, dass nur die neue Variable im Output erscheint.
therapie_long |>
mutate(symptome_p = round(symptome / 7 * 100, digits = 1),
.keep = "none")# A tibble: 200 × 1
symptome_p
<dbl>
1 61.2
2 45.8
3 88.2
4 85.5
5 56.2
6 59.6
7 72.3
8 68.1
9 92.2
10 80.6
# ℹ 190 more rowsacross(): Mehrere Variablen auswählen und verändern
Möchten wir Transformationen auf mehrere Variablen gleichzeitig anwenden, können wir mutate() mit across() verwenden.
Syntax:
across(.cols, .fns, .names)Das erste Argument .cols wählt die Variablen aus, mit denen gearbeitet wird. Das zweite Argument .fns bestimmt eine oder mehrere Funktionen, die auf die ausgewählten Variablen angewendet wird/werden. Mit dem dritten Argument .names kann spezifiziert werden, wie die Variablen im Output benannt werden sollen.
Als Beispiel wollen wir die Symptom-Werte im Datensatz therapie_wide in Prozentwerte umrechnen. Diese Werte befinden sich in den Spalten pretest and posttest. Da diese die einzigen numerischen Variablen im Datensatz sind, können wir zur Auswahl dieser Variablen die Bedingung where(is.numeric) benutzen, die alle numerischen Variablen eines Data Frames auswählt.
Oben haben wir der in mutate() zu bildenden Variable einfach einen Namen gegeben und anschliessend die Formel bzw. Funktion angegeben, mit der die neue Variable erstellt werden soll.
Bei der Bildung/Transformation von mehr als einer Variablen muss zur Definition der Formel/Funktion eine anonymous function erstellt werden, d.h. es muss explizit eine Funktion definiert werden, die auf die ausgewählten Variablen angewendet wird.
Eine anonymous function startet mit function(x) gefolgt von der Definition der Funktion (in diesem Fall eine Kombination von round() und der Formel zur Umrechnung in Prozentwerte), wobei x (das Argument der zu bildenden Funktion) für die jeweils einzufügende Variable steht:
therapie_wide |>
mutate(across(.cols = where(is.numeric),
.fns = function(x) round(x / 7 * 100, digits = 1)))# A tibble: 100 × 4
id gruppe pretest posttest
<fct> <fct> <dbl> <dbl>
1 1 Kontrollgruppe 61.2 45.8
2 2 Kontrollgruppe 88.2 85.5
3 3 Kontrollgruppe 56.2 59.6
4 4 Kontrollgruppe 72.3 68.1
5 5 Kontrollgruppe 92.2 80.6
6 6 Kontrollgruppe 64.1 66.7
7 7 Kontrollgruppe 65.7 60.5
8 8 Kontrollgruppe 63.7 47.8
9 9 Kontrollgruppe 68 58.7
10 10 Kontrollgruppe 73.1 75.6
# ℹ 90 more rowsHier werden die Variablen pretest und posttest direkt verändert/überschrieben.
Mit dem .names-Argument können wir dafür sorgen, dass die transformierten Variablen unter einem neuen Namen dem Datensatz hinzugefügt werden (und die Ausgangsvariablen unverändert im Datensatz verbleiben).
therapie_wide |>
mutate(across(.cols = where(is.numeric),
.fns = function(x) round(x / 7 * 100, digits = 1),
.names = "{.col}_percent"))# A tibble: 100 × 6
id gruppe pretest posttest pretest_percent posttest_percent
<fct> <fct> <dbl> <dbl> <dbl> <dbl>
1 1 Kontrollgruppe 4.29 3.21 61.2 45.8
2 2 Kontrollgruppe 6.18 5.99 88.2 85.5
3 3 Kontrollgruppe 3.93 4.17 56.2 59.6
4 4 Kontrollgruppe 5.06 4.76 72.3 68.1
5 5 Kontrollgruppe 6.45 5.64 92.2 80.6
6 6 Kontrollgruppe 4.49 4.67 64.1 66.7
7 7 Kontrollgruppe 4.60 4.24 65.7 60.5
8 8 Kontrollgruppe 4.46 3.34 63.7 47.8
9 9 Kontrollgruppe 4.76 4.11 68 58.7
10 10 Kontrollgruppe 5.12 5.29 73.1 75.6
# ℹ 90 more rowsIn einer Kurzvariante zur Definition einer anonymous function wird das Wort function durch das Zeichen \ (Backslash) ersetzt. Wir benutzen im Folgenden diese Variante.
Ausserdem können wir die Variablen auch mithilfe eines Vektors von Variablennamen (hier c(pretest, posttest)) auswählen, ohne eine Bedingung wie where(is.numeric) zu verwenden.
Zuletzt müssen die ersten beiden Argumente von across() nicht unbedingt benannt werden (wenn sie in der richtigen Reihenfolge verwendet werden), was den Code noch etwas verkürzt:
therapie_wide |>
mutate(across(c(pretest, posttest), \(x) round(x / 7 * 100, digits = 1),
.names = "{.col}_percent"))# A tibble: 100 × 6
id gruppe pretest posttest pretest_percent posttest_percent
<fct> <fct> <dbl> <dbl> <dbl> <dbl>
1 1 Kontrollgruppe 4.29 3.21 61.2 45.8
2 2 Kontrollgruppe 6.18 5.99 88.2 85.5
3 3 Kontrollgruppe 3.93 4.17 56.2 59.6
4 4 Kontrollgruppe 5.06 4.76 72.3 68.1
5 5 Kontrollgruppe 6.45 5.64 92.2 80.6
6 6 Kontrollgruppe 4.49 4.67 64.1 66.7
7 7 Kontrollgruppe 4.60 4.24 65.7 60.5
8 8 Kontrollgruppe 4.46 3.34 63.7 47.8
9 9 Kontrollgruppe 4.76 4.11 68 58.7
10 10 Kontrollgruppe 5.12 5.29 73.1 75.6
# ℹ 90 more rowsFalls die Funktion, die wir auf mehrere Variablen/Spalten anwenden möchten, kein weiteres Argument hat, kann Sie auch ohne Definition einer anonymous function direkt verwendet werden, dann allerdings ohne Funktionsklammern. Z.B. können wir alle als labelled double eingelesenen Gruppierungsvariablen im Beispieldatensatz mit der Funktion haven::as_factor() zu Faktoren konvertieren und gleichzeitig die eingelesenen Labels zu Faktorstufen machen (vgl. Kap. 3.2).
Für diesen Zweck wurde im Package haven extra eine Funktion namens is.labelled() definiert, mit der wir (zusammen mit across(where())) die als labelled double eingelesenen Variablen identifizieren und auswählen können:
beispieldaten |>
mutate(across(where(is.labelled), haven::as_factor),
.keep = "used")# A tibble: 286 × 6
westost geschlecht bildung_vater bildung_mutter bildung_vater_binaer
<fct> <fct> <fct> <fct> <fct>
1 West weiblich Fachhochschulabschlus… Fachhochschul… hoch
2 West männlich Fachabitur, Abitur Realschulabsc… hoch
3 West weiblich Realschulabschluss (m… Hauptschulabs… niedrig
4 West weiblich Realschulabschluss (m… Fachhochschul… niedrig
5 West weiblich Fachhochschulabschlus… Realschulabsc… hoch
6 West männlich Realschulabschluss (m… Realschulabsc… niedrig
7 West männlich Realschulabschluss (m… Realschulabsc… niedrig
8 West weiblich Fachhochschulabschlus… Fachhochschul… hoch
9 West männlich Realschulabschluss (m… Realschulabsc… niedrig
10 West männlich Fachabitur, Abitur Realschulabsc… hoch
# ℹ 276 more rows
# ℹ 1 more variable: bildung_mutter_binaer <fct>4.4.7 Variablen rekodieren mit case_when()
Manchmal möchte man Variablen nach bestimmten Bedingungen umkodieren, die neue Variable soll also bestimmte Werte aufweisen, je nachdem welche Eigenschaften die Werte der alten Variable haben.
Syntax:
case_when(bedingung_1 ~ "neuer_wert_1",
bedingung_2 ~ "neuer_wert_2")Da man hier Variablen nach bestimmten Bedingungen verändert bzw. neu bildet, wird die Funktion innerhalb von mutate() verwendet, es handelt sich also um eine Art Hilfsfunktion für mutate().
Syntax mit mutate():
mutate(neue_variable = case_when(alte_variable_bedingung_1 ~ "neuer_wert_1",
alte_variable_bedingung_2 ~ "neuer_wert_2")Beispiel
Wir wollen das alter der Jugendlichen im Beispieldatensatz so rekodieren, dass diejenigen, die älter als der Mittelwert sind, den Wert “älter” bekommen, und diejenigen, die jünger als der Mittelwert sind, den Wert “jünger” bekommen.
beispieldaten |>
mutate(alter_dichotom = case_when(alter > mean(alter) ~ "älter",
alter < mean(alter) ~ "jünger"),
.keep = "used")# A tibble: 286 × 2
alter alter_dichotom
<dbl> <chr>
1 13 jünger
2 14 jünger
3 14 jünger
4 14 jünger
5 14 jünger
6 14 jünger
7 14 jünger
8 15 älter
9 15 älter
10 14 jünger
# ℹ 276 more rowsDiese Variable können wir z.B. anschliessend nutzen, um herauszufinden, wie die Altersverteilung in dieser Hinsicht nach Geschlecht aussieht. Dazu müssen wir die Daten aber zuerst wieder zuweisen (hier zu dat_table):
dat_table <- beispieldaten |>
select(alter, geschlecht) |>
drop_na() |>
mutate(geschlecht = haven::as_factor(geschlecht),
alter_dichotom = case_when(alter > mean(alter) ~ "älter",
alter < mean(alter) ~ "jünger"))
table(dat_table$geschlecht, dat_table$alter_dichotom)
älter jünger
männlich 93 59
weiblich 70 64# als Anteile pro Geschlecht (Zeilen der Kreuztabelle)
table(dat_table$geschlecht, dat_table$alter_dichotom) |>
proportions(1) |>
round(2)
älter jünger
männlich 0.61 0.39
weiblich 0.52 0.484.4.8 Faktorstufen rekodieren mit fct_recode()
Um die Stufen eines Faktors umzukodieren, verwenden wir fct_recode() aus dem forcats-Package.
Syntax:
fct_recode(variable,
neuer_wert_1 = "alter_wert_1",
neuer_wert_2 = "alter_wert_2")Auch diese Funktion wird normalerweise innerhalb von mutate() verwendet.
Beispiel
# Kontrollgruppe in "control" und die Therapiegruppe in "treatment" umbenennen
therapie_long |>
mutate(gruppe = fct_recode(gruppe,
control = "Kontrollgruppe",
treatment = "Therapiegruppe"))# A tibble: 200 × 4
id gruppe zeit symptome
<fct> <fct> <fct> <dbl>
1 1 control pretest 4.29
2 1 control posttest 3.21
3 2 control pretest 6.18
4 2 control posttest 5.99
5 3 control pretest 3.93
6 3 control posttest 4.17
7 4 control pretest 5.06
8 4 control posttest 4.76
9 5 control pretest 6.45
10 5 control posttest 5.64
# ℹ 190 more rowsHier wird mit mutate() keine neue Variable gebildet, sondern es wird eine Variable verändert, indem die durch fct_recode() veränderte Variable gruppe der ursprünglichen Variable wiederzugewiesen wird.
Wir können Faktorstufen nicht nur umbenennen, sondern sie auch “zusammenlegen”, um einen Faktor mit weniger Faktorstufen zu erhalten. Das macht man manchmal, wenn manche Kategorien nur sehr geringe Häufigkeiten haben. Z.B. können wir die Altersvariable zunächst als Faktor definieren (es gibt dort nur ganzzahlige Altersangaben):
# Alter als Faktorvariable definieren
beispieldaten <- beispieldaten |>
mutate(alter_f = factor(alter))
table(beispieldaten$alter_f)
13 14 15 16 17
6 117 121 40 2 Es gibt nur sehr wenige 13-Jährige sowie 17-Jährige im Datensatz. Man könnte daher eine dreistufige Altersvariable bilden, bei der diese Alterskategorien mit der jeweils benachbarten zusammengelegt werden:
beispieldaten <- beispieldaten |>
mutate(alter_dreistufig = fct_recode(alter_f,
alt = "17",
alt = "16",
mittel = "15",
jung = "14",
jung = "13"))
table(beispieldaten$alter_dreistufig)
jung mittel alt
123 121 42 4.4.9 Daten gruppieren mit group_by()
Oft wollen wir bestimmte Operationen nicht auf den ganzen Datensatz anwenden, sondern nur auf Subgruppen, welche durch Faktorstufen definiert sind. Dafür gibt es die Funktion group_by() - diese teilt den Datensatz anhand einer Gruppierungsvariable auf, wendet eine Funktion auf jeden Teil/jede Gruppe an, und setzt den Datensatz danach wieder zusammen (split-apply-combine). group_by() wird deshalb meistens in Kombination mit anderen Funktionen verwendet.
Syntax:
df <- group_by(gruppierung_1, gruppierung_2, gruppierung_3)Beispiele
Lassen Sie uns einen neuen Datensatz importieren. In diesem Beispiel wurden 12 Probanden vier Versuchsbedingungen zugeordnet, und als abhängige Variable wurde das Ausmass des aggressiven Verhaltens gemessen. Nehmen wir nun an, dass wir dem Datensatz eine gruppenzentrierte Aggressionsvariable hinzufügen wollen (die Gruppen sind die Bedingungen). Solche gruppenzentrierten Variablen (jede Person hat als Wert die Abweichung vom Gruppenmittelwert der Gruppe, der sie zugehört) werden für bestimmte statistische Verfahren benötigt, die wir in den nächsten Semestern kennenlernen werden.
library(readr)
alk_aggr <- read_csv("data/alkohol-aggression.csv", show_col_types = FALSE)
alk_aggr$alkoholbedingung <- factor(alk_aggr$alkoholbedingung)
alk_aggr# A tibble: 12 × 2
aggressivitaet alkoholbedingung
<dbl> <fct>
1 64 kein_alkohol
2 58 kein_alkohol
3 64 kein_alkohol
4 74 placebo
5 79 placebo
6 72 placebo
7 71 anti_placebo
8 69 anti_placebo
9 67 anti_placebo
10 69 alkohol
11 73 alkohol
12 74 alkohol Wir können group_by() verwenden, um den Datensatz in vier separate Teile zu teilen, berechnen dann den Gruppenmittelwert für jeden einzelnen, und verwenden diesen gleich, um die gruppenzentrierte Variable zu berechnen. Anschliessend werden die Teile wieder “zusammengesetzt” (d.h. es ist wieder ein Datensatz). Da wir das ganze wieder zuweisen, benutzen wir am Ende ungroup().
alk_aggr <- alk_aggr |>
group_by(alkoholbedingung) |>
mutate(group_mean = mean(aggressivitaet),
aggr_c = aggressivitaet - group_mean) |>
ungroup()
alk_aggr# A tibble: 12 × 4
aggressivitaet alkoholbedingung group_mean aggr_c
<dbl> <fct> <dbl> <dbl>
1 64 kein_alkohol 62 2
2 58 kein_alkohol 62 -4
3 64 kein_alkohol 62 2
4 74 placebo 75 -1
5 79 placebo 75 4
6 72 placebo 75 -3
7 71 anti_placebo 69 2
8 69 anti_placebo 69 0
9 67 anti_placebo 69 -2
10 69 alkohol 72 -3
11 73 alkohol 72 1
12 74 alkohol 72 24.4.10 Variablen zusammenfassen mit summarize()
Mit summarize() oder summarise() können wir Variablen zusammenfassen und deskriptive Kennzahlen berechnen. summarize() wird oft zusammen mit group_by() verwendet.
Syntax:
df |> summarize(kennzahl = FUNKTION(variable))FUNKTION ist ein Platzhalter für jede Funktion, die zum Zusammenfassen von Daten verwendet werden kann, z.B. mean() oder sd().
Als Beispiel mit dem therapie_long Datensatz berechnen wir die Mittelwerte und die Standardabweichungen der Variable symptome getrennt für alle Gruppen und Messzeitpunkte:
# Gruppenmittelwerte pro Messzeitpunkt
therapie_long |>
group_by(gruppe, zeit) |>
summarize(Mittelwert = mean(symptome),
Standardabweichung = sd(symptome))`summarise()` has grouped output by 'gruppe'. You can override using the
`.groups` argument.# A tibble: 4 × 4
# Groups: gruppe [2]
gruppe zeit Mittelwert Standardabweichung
<fct> <fct> <dbl> <dbl>
1 Kontrollgruppe pretest 5.06 0.908
2 Kontrollgruppe posttest 4.65 0.834
3 Therapiegruppe pretest 4.82 0.691
4 Therapiegruppe posttest 4.23 0.752Statt wie oben summarize() nur auf eine Variable anzuwenden, können wir diese Funktion mit across() auch auf mehrere Variablen anwenden. Z.B. möchten wir die Mittelwerte der Items swk4 bis swk7 berechnen. Dazu wird die Funktion mean() wieder ohne Funktionsklammern benutzt, da sie innerhalb von across() ohne weiteres Argument verwendet wird:
beispieldaten |>
summarize(across(swk4:swk7, mean))# A tibble: 1 × 4
swk4 swk5 swk6 swk7
<dbl> <dbl> <dbl> <dbl>
1 NA NA NA NAEs scheint NAs zu geben, daher funktioniert mean() nicht ohne das Argument na.rm = TRUE. Damit wir dieses Argument aber innerhalb von across() aufrufen können, müssen wir wieder eine anonymous function definieren:
beispieldaten |>
summarize(across(swk4:swk7, \(x) mean(x, na.rm = TRUE)))# A tibble: 1 × 4
swk4 swk5 swk6 swk7
<dbl> <dbl> <dbl> <dbl>
1 5.01 6.17 6.16 4.97Oder wir löschen die NAs zuerst:
beispieldaten |>
select(swk4:swk7) |>
drop_na() |>
summarize(across(everything(), mean))# A tibble: 1 × 4
swk4 swk5 swk6 swk7
<dbl> <dbl> <dbl> <dbl>
1 5.01 6.17 6.17 4.96Warum unterscheiden sich die Ergebnisse minimal? - Weil oben die NAs pro Variable entfernt wurden, während hier alle Personen gelöscht wurden, die einen fehlenden Wert auf irgendeiner der vier Variablen haben.
4.5 Übungsaufgaben
Jugendliche in West-/Ostdeutschland
library(haven)
beispieldaten <- read_sav("data/beispieldaten.sav")
beispieldaten$westost <- haven::as_factor(beispieldaten$westost)
beispieldaten$geschlecht <- haven::as_factor(beispieldaten$geschlecht)
head(beispieldaten)# A tibble: 6 × 98
ID westost geschlecht alter swk1 swk2 swk3 swk4 swk5 swk6 swk7 swk8
<dbl> <fct> <fct> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 1 West weiblich 13 2 5 6 4 7 5 5 6
2 2 West männlich 14 4 6 4 4 6 5 5 6
3 10 West weiblich 14 5 4 5 6 5 7 7 6
4 11 West weiblich 14 3 6 6 5 7 7 6 6
5 12 West weiblich 14 5 6 6 5 7 6 5 5
6 14 West männlich 14 3 5 5 4 5 7 5 6
# ℹ 86 more variables: swk9 <dbl>, swk10 <dbl>, swk11 <dbl>, swk12 <dbl>,
# swk13 <dbl>, swk14 <dbl>, swk15 <dbl>, swk16 <dbl>, swk17 <dbl>,
# swk18 <dbl>, swk19 <dbl>, swk20 <dbl>, swk21 <dbl>, swk22 <dbl>,
# swk23 <dbl>, swk24 <dbl>, swk25 <dbl>, swk26 <dbl>, swk27 <dbl>,
# swk28 <dbl>, swk29 <dbl>, swk30 <dbl>, swk31 <dbl>, swk32 <dbl>,
# swk33 <dbl>, swk34 <dbl>, swk35 <dbl>, swk36 <dbl>, unteltern1 <dbl>,
# unteltern2 <dbl>, unteltern3 <dbl>, unteltern4 <dbl>, unteltern5 <dbl>, …Durchschnittsalter
Berechnen Sie das mittlere Alter und die Standardabweichung für beide Geschlechter.
Unterstützung durch Freunde und Eltern
Wählen Sie die Variablen unt_freunde (Unterstützung durch Freunde) und unt_eltern (Unterstützung durch Eltern) aus, und machen sie daraus einen long Datensatz. Beide Variablen wurden mit denselben Items gemessen, sie unterscheiden sich lediglich durch die Quelle der Unterstützung (Freunde oder Eltern). Insofern können wir diese Unterstützungsquelle als messwiederholten Faktor auffassen.
Zufriedenheit
Wir wollen nun die Zufriedenheit mit verschiedenen Lebensbereichen untersuchen. Wir brauchen dazu nicht den ganzen Datensatz. Die relevanten Variablen beginnen alle mit leben_, und sollen ausgewählt werden. Die Variable leben_gesamt ist aber schon eine Zusammenfassung der Zufriedenheit mit allen Bereichen, diese wollen wir nicht berücksichtigen.
Stress
Verschiedene Befunde in der Literatur zeigen, dass Mädchen im Jugendalter stärker unter depressiven Verstimmungen leiden als Jungen. Diese Hypothese können wir mit einem t-Test überprüfen, aber bevor wir das tun, können wir uns die mittlere psychische Symptomatik, nach Geschlecht aufgeteilt, deskriptiv anschauen. Berechnen Sie Mittelwert und Standardabweichung der Variablen stress_psychisch für beide Geschlechter getrennt, und runden Sie diese auf drei Nachkommastellen.
Hinweis: Damit wir überhaupt drei Nachkommastellen angezeigt bekommen können, müssen wir die Befehlsfolge immer mit as.data.frame() abschliessen. Der Grund dafür ist, dass der tibble, den die tidyverse-Funktionen erstellen, eine default Printmethode hat, die in manchen Fällen (z.B. in dieser Online-Version des Skripts) automatisch auf zwei Dezimalstellen rundet.