pacman::p_load(tidyverse, ggplot2, ggthemes, psych, haven, EFAutilities, knitr)5 Exploratorische Faktorenanalyse (EFA)
5.1 Setup
Wir lesen die im einführenden Kapitel zur Exploratorischen Datenreduktion vorgestellten Daten zur Lebenszufriedenheit nochmals ein und führen im selben Schritt die Umbenennung und Umsortierung durch:
lifesat <- read_csv("https://github.com/methodenlehre/data/blob/master/statistik_IV/lifesat.csv?raw=true") |>
rename(leben8_familie = leben8,
leben9_familie = leben9,
leben10_familie = leben10,
leben1_schule = leben1,
leben3_schule = leben3,
leben4_schule = leben4,
leben2_selbst = leben2,
leben6_selbst = leben6,
leben5_freunde = leben5,
leben7_freunde = leben7) |>
relocate(leben1_schule, leben3_schule, leben4_schule,
leben2_selbst, leben6_selbst,
leben5_freunde, leben7_freunde,
leben8_familie, leben9_familie, leben10_familie)5.2 Parallelanalyse
Zuerst führen wir eine Parallelanalyse durch. Weil wir die Parallelanalyse jetzt für die Faktorenanalyse berechnen (und nicht wie im vorigen Kapitel für die Hauptkomponentenanalyse), spezifizieren wir jetzt fa = "fa". Ausserdem benutzen wir das Argument fm = "ml", damit die Parallelanalyse auf einer Maximum-Likelihood-Faktorenanalyse basiert.
parallelAnalyse <- fa.parallel(lifesat,
n.iter = 1000, fa = "fa",
fm = "ml", quant = 0.5
)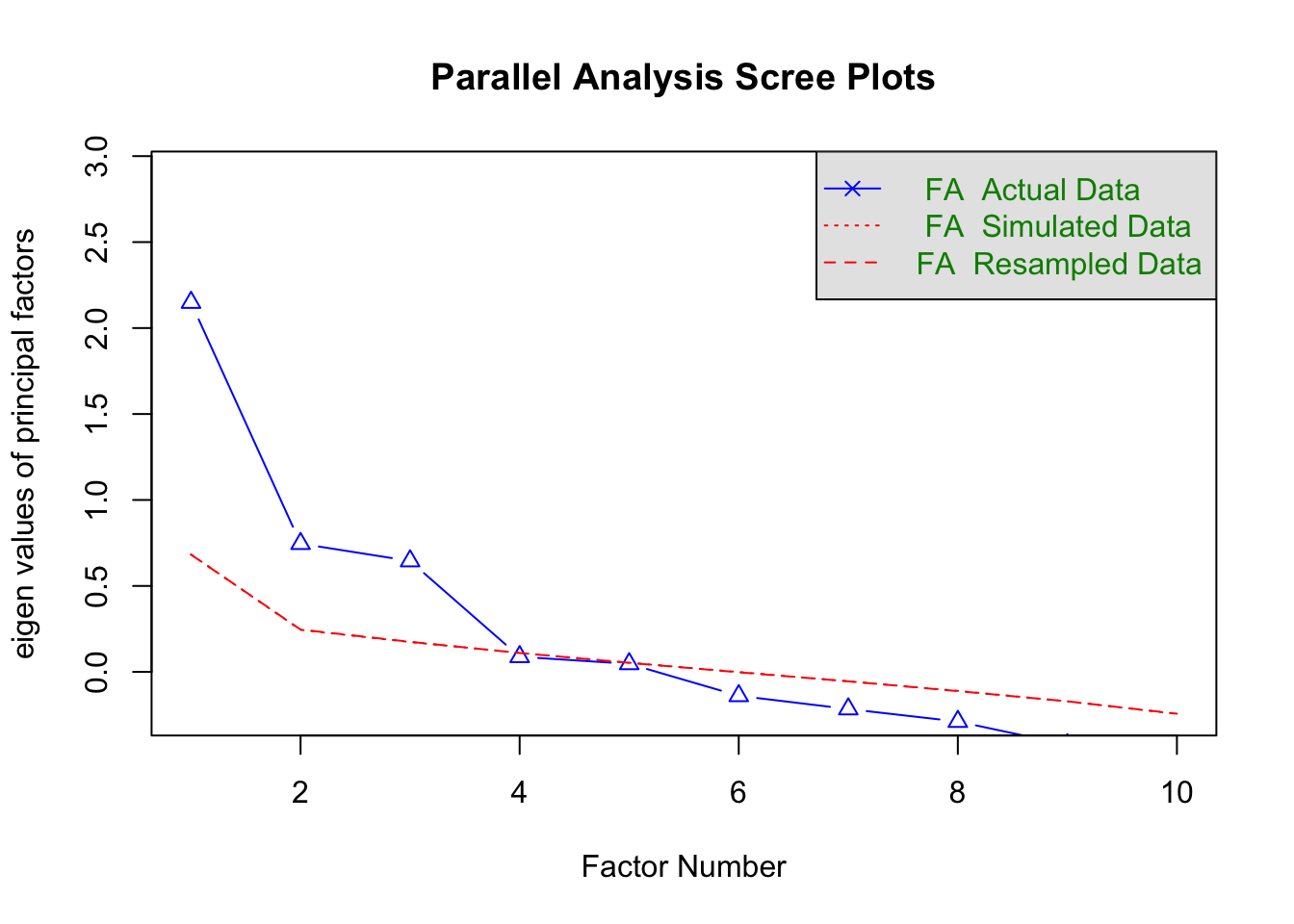
Parallel analysis suggests that the number of factors = 3 and the number of components = NA Die Parallelanalyse zur ML-EFA zeigt, dass auch hier drei Faktoren extrahiert werden sollten. Der Eigenwert des vierten Faktors liegt knapp unter dem Median der 1000 simulierten vierten Eigenwerte einer Zufallsmatrix.
5.3 ML-EFA mit drei unrotierten Faktoren
# Unrotierte EFA mit 3 Faktoren
efa.3.unrotiert <- fa(lifesat, fm = "ml", nfactors = 3, rotate = "none")
print.psych(efa.3.unrotiert, sort = TRUE)Factor Analysis using method = ml
Call: fa(r = lifesat, nfactors = 3, rotate = "none", fm = "ml")
Standardized loadings (pattern matrix) based upon correlation matrix
item ML1 ML2 ML3 h2 u2 com
leben4_schule 3 0.83 -0.42 -0.07 0.87 0.13 1.5
leben3_schule 2 0.55 -0.22 -0.11 0.36 0.64 1.4
leben1_schule 1 0.43 0.00 -0.01 0.18 0.82 1.0
leben10_familie 10 0.41 0.33 0.06 0.28 0.72 2.0
leben9_familie 9 0.48 0.73 -0.20 0.81 0.19 1.9
leben8_familie 8 0.43 0.57 -0.04 0.51 0.49 1.9
leben6_selbst 5 0.27 0.20 0.70 0.60 0.40 1.5
leben2_selbst 4 0.27 0.06 0.59 0.43 0.57 1.4
leben5_freunde 6 0.37 0.14 0.40 0.32 0.68 2.2
leben7_freunde 7 0.23 0.16 0.30 0.17 0.83 2.5
ML1 ML2 ML3
SS loadings 2.09 1.29 1.15
Proportion Var 0.21 0.13 0.12
Cumulative Var 0.21 0.34 0.45
Proportion Explained 0.46 0.28 0.25
Cumulative Proportion 0.46 0.75 1.00
Mean item complexity = 1.7
Test of the hypothesis that 3 factors are sufficient.
df null model = 45 with the objective function = 2.29 with Chi Square = 571.23
df of the model are 18 and the objective function was 0.16
The root mean square of the residuals (RMSR) is 0.04
The df corrected root mean square of the residuals is 0.06
The harmonic n.obs is 255 with the empirical chi square 36.34 with prob < 0.0064
The total n.obs was 255 with Likelihood Chi Square = 39.98 with prob < 0.0021
Tucker Lewis Index of factoring reliability = 0.895
RMSEA index = 0.069 and the 90 % confidence intervals are 0.04 0.098
BIC = -59.76
Fit based upon off diagonal values = 0.97
Measures of factor score adequacy
ML1 ML2 ML3
Correlation of (regression) scores with factors 0.95 0.92 0.84
Multiple R square of scores with factors 0.89 0.84 0.71
Minimum correlation of possible factor scores 0.79 0.68 0.42Insgesamt können mit drei Faktoren 45 % der Gesamtvarianz erklärt werden. Die Eigenwerte entsprechen (anders als bei der PCA) hier übrigens nicht denen der Parallelanalyse. Das hängt damit zusammen, dass die Eigenwerte in der EFA auch von der Anzahl der extrahierten Faktoren abhängen, in der PCA dagegen nicht.
Der Anpassungstest für das Modell (“Test of the hypothesis that 3 factors are sufficient.”) zeigt ein \(\chi^2 = 39.98, p = 0.002\). Somit muss die Nullhypothese, dass drei Faktoren ausreichen, um die Korrelationen zwischen den Items vollständig zu erklären, abgelehnt werden. Auch die weiteren Fit-Indizes, die wir erst bei der CFA genauer kennenlernen werden, zeigen keinen besonders guten Fit an. Aus diesem Grund werden wir weiter unten auch noch eine 4-Faktor-Lösung betrachten.
5.4 ML-EFA mit drei rotierten Faktoren
Zunächst betrachten wir aber eine rotierte Lösung für drei Faktoren. Eine orthogonale Rotation lassen wir diesmal aus und betrachten gleich eine schiefwinklige Rotation. Anders als oben bei der PCA benutzen wir hier keine Oblimin-Rotation, sondern eine oblique Geomin-Rotation. Der Grund ist, dass die Oblimin-Rotation im Package EFAutilities, das uns weiter unten die ML-geschätzten Standardfehler und Konfidenzintervalle der Ladungen und Faktorkorrelationen liefern soll, nicht enthalten ist. Die oblique Geomin-Rotation liefert aber sehr ähnliche Ergebnisse wie eine Oblimin-Rotation.
# Rotierte EFA mit 3 Faktoren
efa.3.rotiert <- fa(lifesat, fm = "ml", nfactors = 3, rotate = "geominQ")
print.psych(efa.3.rotiert, sort = TRUE)Factor Analysis using method = ml
Call: fa(r = lifesat, nfactors = 3, rotate = "geominQ", fm = "ml")
Standardized loadings (pattern matrix) based upon correlation matrix
item ML2 ML3 ML1 h2 u2 com
leben9_familie 9 0.92 -0.06 -0.02 0.81 0.19 1.0
leben8_familie 8 0.69 0.08 0.00 0.51 0.49 1.0
leben10_familie 10 0.43 0.16 0.10 0.28 0.72 1.4
leben6_selbst 5 0.01 0.79 -0.09 0.60 0.40 1.0
leben2_selbst 4 -0.08 0.67 0.03 0.43 0.57 1.0
leben5_freunde 6 0.10 0.49 0.10 0.32 0.68 1.2
leben7_freunde 7 0.11 0.36 0.01 0.17 0.83 1.2
leben4_schule 3 -0.04 0.01 0.94 0.87 0.13 1.0
leben3_schule 2 0.05 -0.05 0.60 0.36 0.64 1.0
leben1_schule 1 0.16 0.06 0.34 0.18 0.82 1.5
ML2 ML3 ML1
SS loadings 1.61 1.52 1.40
Proportion Var 0.16 0.15 0.14
Cumulative Var 0.16 0.31 0.45
Proportion Explained 0.36 0.33 0.31
Cumulative Proportion 0.36 0.69 1.00
With factor correlations of
ML2 ML3 ML1
ML2 1.00 0.28 0.19
ML3 0.28 1.00 0.23
ML1 0.19 0.23 1.00
Mean item complexity = 1.1
Test of the hypothesis that 3 factors are sufficient.
df null model = 45 with the objective function = 2.29 with Chi Square = 571.23
df of the model are 18 and the objective function was 0.16
The root mean square of the residuals (RMSR) is 0.04
The df corrected root mean square of the residuals is 0.06
The harmonic n.obs is 255 with the empirical chi square 36.34 with prob < 0.0064
The total n.obs was 255 with Likelihood Chi Square = 39.98 with prob < 0.0021
Tucker Lewis Index of factoring reliability = 0.895
RMSEA index = 0.069 and the 90 % confidence intervals are 0.04 0.098
BIC = -59.76
Fit based upon off diagonal values = 0.97
Measures of factor score adequacy
ML2 ML3 ML1
Correlation of (regression) scores with factors 0.92 0.87 0.94
Multiple R square of scores with factors 0.86 0.75 0.88
Minimum correlation of possible factor scores 0.71 0.50 0.77Wie bei der PCA ergeben sich hier ein Familien-Faktor (ML2), eine Selbst- und Freunde-Faktor (ML3) und ein Schul-Faktor (ML1). Die Faktorkorrelationen sind ähnlich wie bei der PCA relativ gleichmässig: \((r_{ML_1ML_2} = 0.19, r_{ML_1ML_3} = 0.23, r_{ML_2ML_3} = 0.28)\).
5.4.1 Konfidenzintervalle und Standardfehler
Mit der Funktion fa() aus psych ist es nicht möglich, Standardfehler und Konfidenzintervalle für die Ladungen einer Maximum-Likelihood-EFA zu berechnen. Mit der Funktion efa() aus dem Package EFAutilities ist dies dagegen möglich. Wie oben werden mit der ML-Methode (fm = "ml") 3 Faktoren (factors = 3) extrahiert und dann eine oblique Geomin-Rotation durchgeführt (rtype = "oblique", rotation = "geomin"). Ausserdem werden mit LConfid = c(.95, .90) zwei Konfidenzkoeffizienten spezifiziert: der erste (0.95) bezieht sich auf die Konfidenzintervalle der Faktorladungen und -korrelationen, der zweite (0.90) ausschliesslich auf den Fit-Index RMSEA, den wir erst beim Thema CFA genauer betrachten. Alle uns hier interessierenden Konfidenzintervalle sind also 95 %-Konfidenzintervalle.
Da der Output von EFAutilities::efa() unübersichtlich ist und wir uns hier nur für das Feature “Standardfehler und CIs” der Ladungen interessieren, haben wir den Output so formatiert, dass eine Tabelle mit den Ladungen zusammen mit ihren Standardfehlern (SEs) sowie eine weitere mit den Ladungen zusammen mit ihren Konfidenzintervallen (CIs) ausgegeben wird:
efa.ci.rotated <- EFAutilities::efa(lifesat,
fm = "ml", factors = 3,
rtype = "oblique",
rotation = "geomin",
LConfid = c(.95, .90)
)| F1 | F2 | F3 | |
|---|---|---|---|
| leben1_schule | 0.34 [se=0.07] | 0.16 [se=0.08] | 0.06 [se=0.07] |
| leben3_schule | 0.6 [se=0.09] | 0.05 [se=0.06] | -0.05 [se=0.06] |
| leben4_schule | 0.94 [se=0.11] | -0.04 [se=0.02] | 0.01 [se=0.02] |
| leben2_selbst | 0.03 [se=0.04] | -0.08 [se=0.06] | 0.67 [se=0.06] |
| leben6_selbst | -0.09 [se=0.05] | 0.01 [se=0.02] | 0.79 [se=0.06] |
| leben5_freunde | 0.1 [se=0.06] | 0.1 [se=0.07] | 0.49 [se=0.08] |
| leben7_freunde | 0.01 [se=0.06] | 0.11 [se=0.08] | 0.36 [se=0.09] |
| leben8_familie | 0 [se=0.04] | 0.69 [se=0.07] | 0.08 [se=0.06] |
| leben9_familie | -0.02 [se=0.03] | 0.92 [se=0.07] | -0.06 [se=0.03] |
| leben10_familie | 0.1 [se=0.06] | 0.43 [se=0.06] | 0.16 [se=0.07] |
| F1 | F2 | F3 | |
|---|---|---|---|
| leben1_schule | 0.34 [0.197; 0.484] | 0.16 [0.009; 0.319] | 0.06 [-0.087; 0.199] |
| leben3_schule | 0.6 [0.418; 0.777] | 0.05 [-0.056; 0.162] | -0.05 [-0.167; 0.063] |
| leben4_schule | 0.94 [0.725; 1.148] | -0.04 [-0.081; 0.007] | 0.01 [-0.035; 0.059] |
| leben2_selbst | 0.03 [-0.039; 0.099] | -0.08 [-0.195; 0.027] | 0.67 [0.552; 0.785] |
| leben6_selbst | -0.09 [-0.195; 0.013] | 0.01 [-0.032; 0.043] | 0.79 [0.666; 0.914] |
| leben5_freunde | 0.1 [-0.018; 0.226] | 0.1 [-0.039; 0.237] | 0.49 [0.331; 0.652] |
| leben7_freunde | 0.01 [-0.12; 0.133] | 0.11 [-0.043; 0.263] | 0.36 [0.192; 0.532] |
| leben8_familie | 0 [-0.078; 0.069] | 0.69 [0.548; 0.833] | 0.08 [-0.044; 0.197] |
| leben9_familie | -0.02 [-0.07; 0.032] | 0.92 [0.788; 1.052] | -0.06 [-0.114; -0.01] |
| leben10_familie | 0.1 [-0.019; 0.224] | 0.43 [0.307; 0.545] | 0.16 [0.013; 0.303] |
\(~\)
In beiden Tabelle ist jetzt F1 der Schul-Faktor, F2 der Familien-Faktor, und F3 der Selbst- und Freunde-Faktor. Die Konfidenzintervalle aller Ladungen von Items auf einem zugehörigen Faktor sollten den Wert 0 nicht beinhalten und damit signifikant sein, während die Konfidenzintervalle aller Ladungen von Items auf einem nicht zugehörigen Faktor den Wert 0 möglichst beinhalten sollten (nicht signifikante Querladungen).
Das ist auch weitgehend der Fall: Ausnahmen sind nur die (Quer-)ladungen von leben1_schule auf F2 (Familie), leben10_familie auf F3 (Selbst und Freunde) sowie die sehr kleine (und negative) Querladung von leben9_familie auf F3 (Selbst und Freunde). Die CIs dieser Ladungen beinhalten den Wert 0 nicht, sind daher also als signifikant zu werten. Da sie aber kleiner als |0.30| sind, würden wir sie trotzdem nicht als problematische Querladungen identifizieren, die die Einfachstruktur in Frage stellen.
Die folgende Tabelle stellt die Faktorkorrelationen mit den zugehörigen Konfidenzintervallen dar. Alle Faktorkorrelationen sind signifikant, da das CI den Wert 0 jeweils nicht enthält.
Anmerkung: Wegen der Darstellung in Matrixform sind 1) alle Korrelationen der Faktoren F1, F2, und F3 untereinander doppelt enthalten; sowie 2) die Korrelationen der Faktoren mit sich selbst enthalten (alle gleich 1, mit CIs [1; 1]).
| F1 | F2 | F3 | |
|---|---|---|---|
| F1 | 1 [1; 1] | 0.19 [0.052; 0.329] | 0.23 [0.083; 0.375] |
| F2 | 0.19 [0.052; 0.329] | 1 [1; 1] | 0.28 [0.13; 0.412] |
| F3 | 0.23 [0.083; 0.375] | 0.28 [0.13; 0.412] | 1 [1; 1] |
Die direkte Ausgabe der Standardfehler sowie der Unter- und Obergrenzen der Konfidenzintervalle der Faktorladungen und -korrelationen erhält man folgendermassen:
efa.ci.rotated$rotatedse # Standardfehler der rotierten Ladungen F1 F2 F3
MV1 0.07322802 0.07896103 0.07279361
MV2 0.09158024 0.05556916 0.05878619
MV3 0.10781066 0.02245987 0.02412222
MV4 0.03522472 0.05666011 0.05938846
MV5 0.05311117 0.01920808 0.06309384
MV6 0.06231455 0.07030520 0.08196281
MV7 0.06442080 0.07815180 0.08697361
MV8 0.03744344 0.07262921 0.06157801
MV9 0.02606300 0.06726595 0.02650508
MV10 0.06193935 0.06048521 0.07405747efa.ci.rotated$rotatedlow # Untere Grenzen CIs der rotierten Ladungen F1 F2 F3
MV1 0.19675620 0.009496762 -0.08670164
MV2 0.41832842 -0.056311535 -0.16718701
MV3 0.72500289 -0.081480901 -0.03539109
MV4 -0.03939967 -0.194650246 0.55190710
MV5 -0.19524398 -0.031849243 0.66624057
MV6 -0.01838276 -0.038735630 0.33080106
MV7 -0.11986351 -0.043243222 0.19155951
MV8 -0.07784889 0.548353710 -0.04430300
MV9 -0.06986992 0.788387740 -0.11380312
MV10 -0.01853277 0.307441365 0.01287590efa.ci.rotated$rotatedupper # Obere Grenzen CIs der rotierten Ladungen F1 F2 F3
MV1 0.48380477 0.31901832 0.198644065
MV2 0.77731634 0.16151558 0.063250623
MV3 1.14761292 0.00656017 0.059166261
MV4 0.09867869 0.02745330 0.784705589
MV5 0.01294800 0.04344503 0.913563882
MV6 0.22588577 0.23685571 0.652089373
MV7 0.13266140 0.26310622 0.532489811
MV8 0.06892669 0.83305499 0.197078345
MV9 0.03229516 1.05206541 -0.009905117
MV10 0.22426502 0.54453905 0.303175858efa.ci.rotated$Philow # Untere Grenzen CIs der Faktorkorrelationen F1 F2 F3
F1 1.00000000 0.0517378 0.08306656
F2 0.05173780 1.0000000 0.13040290
F3 0.08306656 0.1304029 1.00000000efa.ci.rotated$Phiupper # Obere Grenzen CIs der Faktorkorrelationen F1 F2 F3
F1 1.0000000 0.3293467 0.3748064
F2 0.3293467 1.0000000 0.4117469
F3 0.3748064 0.4117469 1.0000000Visualisierung:
Welche Items laden auf welche Faktoren? Hier sieht man auch die Korrelationen zwischen den Faktoren:
fa.diagram(efa.3.rotiert, cut = 0)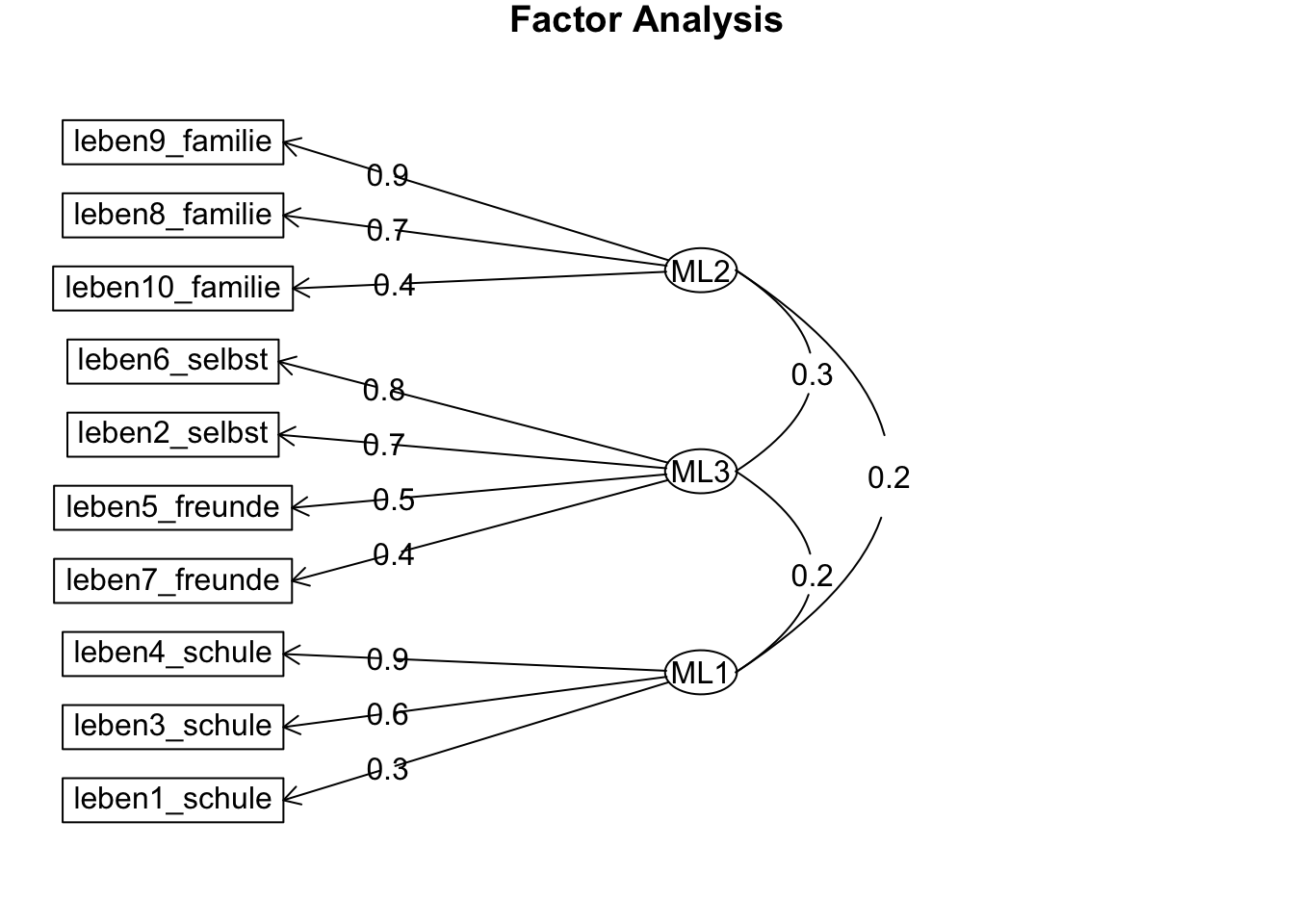
5.5 ML-EFA mit vier unrotierten Faktoren
Unrotierte exploratorische Faktorenanalyse mit 4 Faktoren:
efa.4.unrotiert <- fa(lifesat, fm = "ml", nfactors = 4, rotate = "none")
print.psych(efa.4.unrotiert, sort = TRUE)Factor Analysis using method = ml
Call: fa(r = lifesat, nfactors = 4, rotate = "none", fm = "ml")
Standardized loadings (pattern matrix) based upon correlation matrix
item ML1 ML3 ML4 ML2 h2 u2 com
leben5_freunde 6 0.83 -0.01 0.00 0.56 1.00 0.005 1.8
leben6_selbst 5 0.83 0.00 0.00 -0.56 1.00 0.005 1.8
leben2_selbst 4 0.52 0.01 0.11 -0.16 0.31 0.687 1.3
leben7_freunde 7 0.42 0.07 -0.04 0.13 0.20 0.799 1.3
leben9_familie 9 0.21 0.77 -0.42 0.06 0.81 0.188 1.7
leben8_familie 8 0.25 0.60 -0.29 -0.01 0.51 0.492 1.8
leben10_familie 10 0.28 0.44 -0.09 -0.03 0.28 0.723 1.8
leben1_schule 1 0.19 0.33 0.21 0.03 0.19 0.808 2.4
leben4_schule 3 0.19 0.47 0.73 0.12 0.80 0.198 2.0
leben3_schule 2 0.06 0.40 0.48 0.06 0.39 0.605 2.0
ML1 ML3 ML4 ML2
SS loadings 2.08 1.64 1.08 0.69
Proportion Var 0.21 0.16 0.11 0.07
Cumulative Var 0.21 0.37 0.48 0.55
Proportion Explained 0.38 0.30 0.20 0.13
Cumulative Proportion 0.38 0.68 0.87 1.00
Mean item complexity = 1.8
Test of the hypothesis that 4 factors are sufficient.
df null model = 45 with the objective function = 2.29 with Chi Square = 571.23
df of the model are 11 and the objective function was 0.06
The root mean square of the residuals (RMSR) is 0.02
The df corrected root mean square of the residuals is 0.05
The harmonic n.obs is 255 with the empirical chi square 12.53 with prob < 0.33
The total n.obs was 255 with Likelihood Chi Square = 14.32 with prob < 0.22
Tucker Lewis Index of factoring reliability = 0.974
RMSEA index = 0.034 and the 90 % confidence intervals are 0 0.079
BIC = -46.63
Fit based upon off diagonal values = 0.99
Measures of factor score adequacy
ML1 ML3 ML4 ML2
Correlation of (regression) scores with factors 1.00 0.92 0.90 1.00
Multiple R square of scores with factors 1.00 0.85 0.81 0.99
Minimum correlation of possible factor scores 0.99 0.70 0.62 0.98Insgesamt können mit vier Faktoren 55 % der Gesamtvarianz erklärt werden. Der Anpassungstest für das Modell (“Test of the hypothesis that 4 factors are sufficient.”) zeigt ein \(\chi^2 = 14.32, p = 0.22\). Somit kann die Nullhypothese, dass vier Faktoren ausreichen, beibehalten werden. Auch die weiteren Fit-Indizes sind deutlich besser als bei der 3-Faktor-Lösung.
5.5.1 ML-EFA mit vier rotierten Faktoren
Geomin-rotierte exploratorische Faktorenanalyse mit 4 Faktoren:
efa.4.rotiert <- fa(lifesat, fm = "ml", nfactors = 4, rotate = "geominQ")
print.psych(efa.4.rotiert, sort = TRUE)Factor Analysis using method = ml
Call: fa(r = lifesat, nfactors = 4, rotate = "geominQ", fm = "ml")
Standardized loadings (pattern matrix) based upon correlation matrix
item ML3 ML4 ML1 ML2 h2 u2 com
leben9_familie 9 0.92 -0.02 -0.07 0.00 0.81 0.188 1.0
leben8_familie 8 0.70 0.01 0.05 0.00 0.51 0.492 1.0
leben10_familie 10 0.43 0.12 0.13 0.02 0.28 0.723 1.4
leben4_schule 3 -0.04 0.90 0.00 0.03 0.80 0.198 1.0
leben3_schule 2 0.05 0.63 -0.04 -0.06 0.39 0.605 1.0
leben1_schule 1 0.16 0.35 0.06 0.03 0.19 0.808 1.5
leben6_selbst 5 0.02 -0.03 1.00 -0.02 1.00 0.005 1.0
leben2_selbst 4 -0.04 0.09 0.47 0.15 0.31 0.687 1.3
leben5_freunde 6 0.00 0.00 -0.01 1.00 1.00 0.005 1.0
leben7_freunde 7 0.09 -0.01 0.12 0.35 0.20 0.799 1.4
ML3 ML4 ML1 ML2
SS loadings 1.59 1.37 1.32 1.20
Proportion Var 0.16 0.14 0.13 0.12
Cumulative Var 0.16 0.30 0.43 0.55
Proportion Explained 0.29 0.25 0.24 0.22
Cumulative Proportion 0.29 0.54 0.78 1.00
With factor correlations of
ML3 ML4 ML1 ML2
ML3 1.00 0.20 0.21 0.25
ML4 0.20 1.00 0.13 0.23
ML1 0.21 0.13 1.00 0.40
ML2 0.25 0.23 0.40 1.00
Mean item complexity = 1.2
Test of the hypothesis that 4 factors are sufficient.
df null model = 45 with the objective function = 2.29 with Chi Square = 571.23
df of the model are 11 and the objective function was 0.06
The root mean square of the residuals (RMSR) is 0.02
The df corrected root mean square of the residuals is 0.05
The harmonic n.obs is 255 with the empirical chi square 12.53 with prob < 0.33
The total n.obs was 255 with Likelihood Chi Square = 14.32 with prob < 0.22
Tucker Lewis Index of factoring reliability = 0.974
RMSEA index = 0.034 and the 90 % confidence intervals are 0 0.079
BIC = -46.63
Fit based upon off diagonal values = 0.99
Measures of factor score adequacy
ML3 ML4 ML1 ML2
Correlation of (regression) scores with factors 0.92 0.91 1.00 1.00
Multiple R square of scores with factors 0.85 0.83 0.99 1.00
Minimum correlation of possible factor scores 0.71 0.67 0.99 0.99Die Lösung zeigt eine klare Einfachstruktur. Keine Ladung auf einem nicht-zugehörigen Faktor ist > 0.16. Die Einfachstruktur deckt sich auch mit der inhaltlichen Ausrichtung der Lebenszufriedenheitsbereiche: jetzt haben alle Bereiche (Familie, Schule, Selbst, Freunde) ihren eigenen Faktor.
Die Faktorkorrelationen der vier Faktoren sind jetzt nicht mehr so gleichmässig wie noch bei der 3-Komponenten-Lösung: die grösste Korrelation zeigt sich zwischen dem Selbst- und dem Freunde-Faktor: \(r_{ML_1ML_2} = 0.40\) und die geringste zwischen dem Selbst- und dem Schul-Faktor: \(r_{ML_1ML_4} = 0.13\).
Auf die zusätzliche Berechnung von Standardfehlern und Konfidenzintervallen mit EFAutilities::efa() verzichten wir hier.
Visualisierung:
Faktorladung je Item graphisch dargestellt:
factor.plot(efa.4.rotiert)
\(~\)
Welche Items laden auf welche Faktoren?
fa.diagram(efa.4.rotiert, cut = 0)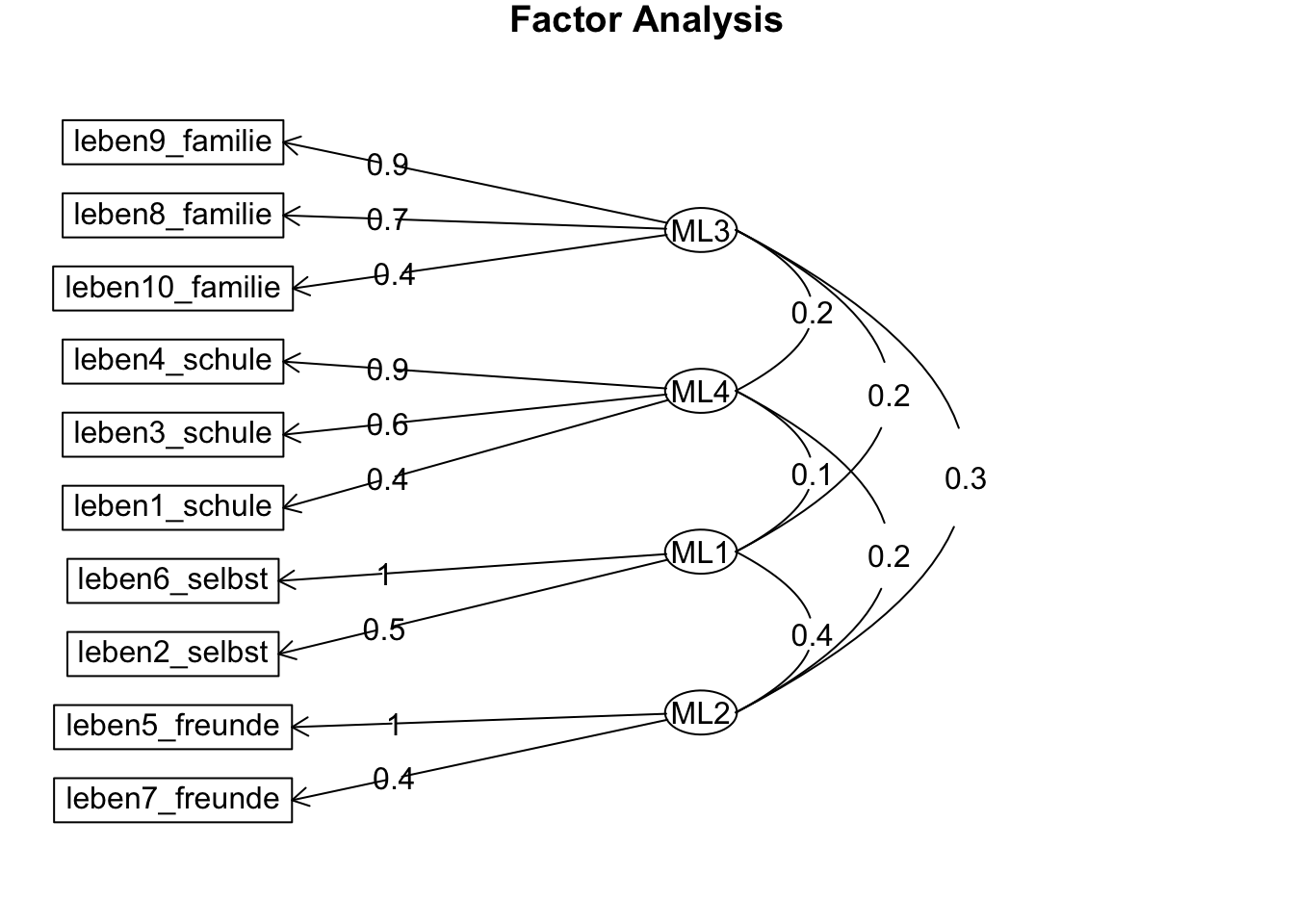
5.6 Zusammenfassung ML-EFA
In der ML-EFA zeigten sich laut Parallelanalyse auch drei Faktoren. Allerdings ist der Chi-Quadrat-Test zur Frage, ob drei Faktoren ausreichen, signifikant. Dies spricht für eine 4-Faktor-Lösung, bei der sich der Selbst-Freunde-Faktor in zwei Faktoren aufspaltet, die die Inhaltsbereiche Selbst und Freunde repräsentieren. Obwohl sich Selbst- und Freunde-Items also viel Varianz teilen und die Parallelanalyse deren Gruppierung auf einem Faktor bevorzugt, müssen diese Inhaltsbereiche im Sinne einer vollständigen Repräsentation der Zusammenhänge separat betrachtet werden.